+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12866 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Nog1-TAP associated immature ribosomal particle population A from S. cerevisiae | |||||||||
 マップデータ マップデータ | Nog1-TAP associated ribosomal particle population A from S. cerevisiae | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ribosomal assembly state / RIBOSOME | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of ribosomal subunit export from nucleus / positive regulation of ATP-dependent activity / maturation of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / pre-mRNA 5'-splice site binding / maturation of 5.8S rRNA / cleavage in ITS2 between 5.8S rRNA and LSU-rRNA of tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / ribosomal large subunit binding / SRP-dependent cotranslational protein targeting to membrane / GTP hydrolysis and joining of the 60S ribosomal subunit / negative regulation of mRNA splicing, via spliceosome ...regulation of ribosomal subunit export from nucleus / positive regulation of ATP-dependent activity / maturation of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / pre-mRNA 5'-splice site binding / maturation of 5.8S rRNA / cleavage in ITS2 between 5.8S rRNA and LSU-rRNA of tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / ribosomal large subunit binding / SRP-dependent cotranslational protein targeting to membrane / GTP hydrolysis and joining of the 60S ribosomal subunit / negative regulation of mRNA splicing, via spliceosome / preribosome, large subunit precursor / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / ATPase activator activity / Formation of a pool of free 40S subunits / nuclear-transcribed mRNA catabolic process / L13a-mediated translational silencing of Ceruloplasmin expression / translational elongation / ribosomal large subunit export from nucleus / regulation of translational fidelity / protein-RNA complex assembly / ribosomal subunit export from nucleus / maturation of LSU-rRNA / translation initiation factor activity / assembly of large subunit precursor of preribosome / ribosomal large subunit biogenesis / maturation of LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / cytosolic ribosome assembly / maturation of SSU-rRNA / macroautophagy / small-subunit processome / maintenance of translational fidelity / rRNA processing / ribosome biogenesis / ATPase binding / ribosomal large subunit assembly / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / cytoplasmic translation / negative regulation of translation / rRNA binding / structural constituent of ribosome / ribosome / translation / GTPase activity / mRNA binding / GTP binding / nucleolus / RNA binding / zinc ion binding / nucleoplasm / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |    | |||||||||
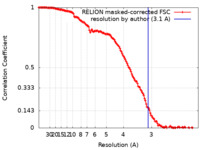
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Milkereit P / Poell G | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2021 ジャーナル: PLoS One / 年: 2021タイトル: Analysis of subunit folding contribution of three yeast large ribosomal subunit proteins required for stabilisation and processing of intermediate nuclear rRNA precursors. 著者: Gisela Pöll / Michael Pilsl / Joachim Griesenbeck / Herbert Tschochner / Philipp Milkereit /  要旨: In yeast and human cells many of the ribosomal proteins (r-proteins) are required for the stabilisation and productive processing of rRNA precursors. Functional coupling of r-protein assembly with ...In yeast and human cells many of the ribosomal proteins (r-proteins) are required for the stabilisation and productive processing of rRNA precursors. Functional coupling of r-protein assembly with the stabilisation and maturation of subunit precursors potentially promotes the production of ribosomes with defined composition. To further decipher mechanisms of such an intrinsic quality control pathway we analysed here the contribution of three yeast large ribosomal subunit r-proteins rpL2 (uL2), rpL25 (uL23) and rpL34 (eL34) for intermediate nuclear subunit folding steps. Structure models obtained from single particle cryo-electron microscopy analyses provided evidence for specific and hierarchic effects on the stable positioning and remodelling of large ribosomal subunit domains. Based on these structural and previous biochemical data we discuss possible mechanisms of r-protein dependent hierarchic domain arrangement and the resulting impact on the stability of misassembled subunits. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12866.map.gz emd_12866.map.gz | 192.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12866-v30.xml emd-12866-v30.xml emd-12866.xml emd-12866.xml | 65.5 KB 65.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12866_fsc.xml emd_12866_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12866.png emd_12866.png | 144.5 KB | ||
| Filedesc metadata |  emd-12866.cif.gz emd-12866.cif.gz | 13.6 KB | ||
| その他 |  emd_12866_half_map_1.map.gz emd_12866_half_map_1.map.gz emd_12866_half_map_2.map.gz emd_12866_half_map_2.map.gz | 194.3 MB 194.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12866 http://ftp.pdbj.org/pub/emdb/structures/EMD-12866 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12866 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12866 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12866_validation.pdf.gz emd_12866_validation.pdf.gz | 1002.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12866_full_validation.pdf.gz emd_12866_full_validation.pdf.gz | 1001.9 KB | 表示 | |
| XML形式データ |  emd_12866_validation.xml.gz emd_12866_validation.xml.gz | 21.7 KB | 表示 | |
| CIF形式データ |  emd_12866_validation.cif.gz emd_12866_validation.cif.gz | 28.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12866 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12866 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12866 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12866 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7of1MC  7oh3C  7ohpC  7ohqC  7ohrC  7ohsC  7ohtC  7ohuC  7ohvC  7ohwC  7ohxC  7ohyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10776 (タイトル: Nog1 TAP associated immature ribosomal particles from S. cerevisiae EMPIAR-10776 (タイトル: Nog1 TAP associated immature ribosomal particles from S. cerevisiaeData size: 8.3 TB Data #1: Unaligned multiframe micrographs of Nog1-TAP associated immature ribosomal particles from S. cerevisisae [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12866.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12866.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nog1-TAP associated ribosomal particle population A from S. cerevisiae | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0635 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
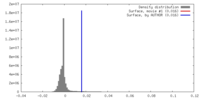
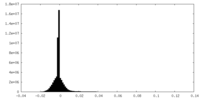
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Nog1-TAP associated ribosomal particle population A from S....
| ファイル | emd_12866_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nog1-TAP associated ribosomal particle population A from S. cerevisiae half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
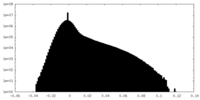
| 密度ヒストグラム |
-ハーフマップ: Nog1-TAP associated ribosomal particle population A from S....
| ファイル | emd_12866_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nog1-TAP associated ribosomal particle population A from S. cerevisiae half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
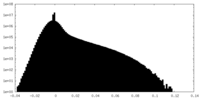
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Nog1-TAP associated immature ribosomal particles.
+超分子 #1: Nog1-TAP associated immature ribosomal particles.
+分子 #1: 25S rRNA
+分子 #2: 5.8S rRNA
+分子 #3: 5S rRNA
+分子 #4: 60S ribosomal protein L2-A
+分子 #5: 60S ribosomal protein L3
+分子 #6: 60S ribosomal protein L4-A
+分子 #7: 60S ribosomal protein L6-A
+分子 #8: 60S ribosomal protein L7-A
+分子 #9: 60S ribosomal protein L8-A
+分子 #10: 60S ribosomal protein L9-A
+分子 #11: 60S ribosomal protein L13-A
+分子 #12: 60S ribosomal protein L14-A
+分子 #13: 60S ribosomal protein L15-A
+分子 #14: 60S ribosomal protein L16-A
+分子 #15: 60S ribosomal protein L17-A
+分子 #16: 60S ribosomal protein L18-A
+分子 #17: 60S ribosomal protein L19-A
+分子 #18: 60S ribosomal protein L20-A
+分子 #19: 60S ribosomal protein L21-A
+分子 #20: 60S ribosomal protein L22-A
+分子 #21: 60S ribosomal protein L23-A
+分子 #22: Ribosome assembly factor MRT4
+分子 #23: 60S ribosomal protein L25
+分子 #24: 60S ribosomal protein L26-A
+分子 #25: 60S ribosomal protein L27-A
+分子 #26: 60S ribosomal protein L28
+分子 #27: Nucleolar GTP-binding protein 1
+分子 #28: 60S ribosomal protein L30
+分子 #29: 60S ribosomal protein L31-A
+分子 #30: 60S ribosomal protein L32
+分子 #31: 60S ribosomal protein L33-A
+分子 #32: 60S ribosomal protein L34-A
+分子 #33: 60S ribosomal protein L35-A
+分子 #34: 60S ribosomal protein L36-A
+分子 #35: 60S ribosomal protein L37-A
+分子 #36: 60S ribosomal protein L38
+分子 #37: 60S ribosomal protein L39
+分子 #38: Nucleolar GTP-binding protein 2
+分子 #39: 60S ribosomal protein L43-A
+分子 #40: Ribosome biogenesis protein NSA2
+分子 #41: Ribosome biogenesis protein RLP24
+分子 #42: Eukaryotic translation initiation factor 6
+分子 #43: MAGNESIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 200 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.4 kPa | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均露光時間: 5.16 sec. / 平均電子線量: 84.67 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-7of1: |
 ムービー
ムービー コントローラー
コントローラー


































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)