+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12864 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle reconstruction of open pentamer of the Haliangium ochraceum encapsulin from symmetry expansion of icosahedral single particle reconstruction | |||||||||
 マップデータ マップデータ | Post process map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Encapsulin / encapsulated ferritin / haliangium ochraceum / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報encapsulin nanocompartment / ferroxidase / ferroxidase activity / iron ion transport / intracellular iron ion homeostasis / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Haliangium ochraceum (バクテリア) / Haliangium ochraceum (バクテリア) /  Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) | |||||||||
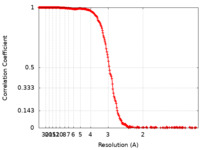
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.64 Å | |||||||||
 データ登録者 データ登録者 | Marles-Wright J / Basle A | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Pore dynamics and asymmetric cargo loading in an encapsulin nanocompartment. 著者: Jennifer Ross / Zak McIver / Thomas Lambert / Cecilia Piergentili / Jasmine Emma Bird / Kelly J Gallagher / Faye L Cruickshank / Patrick James / Efrain Zarazúa-Arvizu / Louise E Horsfall / ...著者: Jennifer Ross / Zak McIver / Thomas Lambert / Cecilia Piergentili / Jasmine Emma Bird / Kelly J Gallagher / Faye L Cruickshank / Patrick James / Efrain Zarazúa-Arvizu / Louise E Horsfall / Kevin J Waldron / Marcus D Wilson / C Logan Mackay / Arnaud Baslé / David J Clarke / Jon Marles-Wright /  要旨: Encapsulins are protein nanocompartments that house various cargo enzymes, including a family of decameric ferritin-like proteins. Here, we study a recombinant encapsulin:encapsulated ferritin ...Encapsulins are protein nanocompartments that house various cargo enzymes, including a family of decameric ferritin-like proteins. Here, we study a recombinant encapsulin:encapsulated ferritin complex using cryo-electron microscopy and hydrogen/deuterium exchange mass spectrometry to gain insight into the structural relationship between the encapsulin shell and its protein cargo. An asymmetric single-particle reconstruction reveals four encapsulated ferritin decamers in a tetrahedral arrangement within the encapsulin nanocompartment. This leads to a symmetry mismatch between the protein cargo and the icosahedral encapsulin shell. The encapsulated ferritin decamers are offset from the interior face of the encapsulin shell. Using hydrogen/deuterium exchange mass spectrometry, we observed the dynamic behavior of the major fivefold pore in the encapsulin shell and show the pore opening via the movement of the encapsulin A-domain. These data will accelerate efforts to engineer the encapsulation of heterologous cargo proteins and to alter the permeability of the encapsulin shell via pore modifications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12864.map.gz emd_12864.map.gz | 26.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12864-v30.xml emd-12864-v30.xml emd-12864.xml emd-12864.xml | 23.2 KB 23.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12864_fsc.xml emd_12864_fsc.xml | 26.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12864.png emd_12864.png | 87.4 KB | ||
| マスクデータ |  emd_12864_msk_1.map emd_12864_msk_1.map | 729 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12864.cif.gz emd-12864.cif.gz | 6.7 KB | ||
| その他 |  emd_12864_additional_1.map.gz emd_12864_additional_1.map.gz emd_12864_half_map_1.map.gz emd_12864_half_map_1.map.gz emd_12864_half_map_2.map.gz emd_12864_half_map_2.map.gz | 577 MB 583.8 MB 583.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12864 http://ftp.pdbj.org/pub/emdb/structures/EMD-12864 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12864 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12864 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7oeuMC  7odwC  7oe2C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10716 (タイトル: Cryo electron microscopy of single particles of the Haliangium ochraceum Encapsulin:encapsulated ferritin encapsulin nano compartment EMPIAR-10716 (タイトル: Cryo electron microscopy of single particles of the Haliangium ochraceum Encapsulin:encapsulated ferritin encapsulin nano compartmentData size: 1.7 TB Data #1: Multi-frame micrographs and particle sets for Haliangium ochraceum encapsulin:encapsulated ferritin reconstruction [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12864.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12864.map.gz / 形式: CCP4 / 大きさ: 729 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post process map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
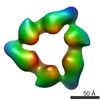
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.652 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12864_msk_1.map emd_12864_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
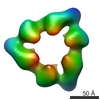
| 投影像・断面図 |
| ||||||||||||
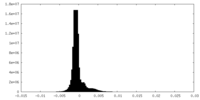
| 密度ヒストグラム |
-追加マップ: Refine 3D map
| ファイル | emd_12864_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refine 3D map | ||||||||||||
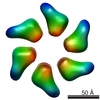
| 投影像・断面図 |
| ||||||||||||
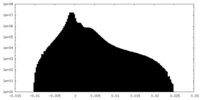
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_12864_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
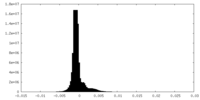
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_12864_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
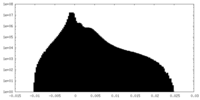
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of Haliangium ochraceum encapsulin and encapsulat...
| 全体 | 名称: Ternary complex of Haliangium ochraceum encapsulin and encapsulated ferritin proteins |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of Haliangium ochraceum encapsulin and encapsulat...
| 超分子 | 名称: Ternary complex of Haliangium ochraceum encapsulin and encapsulated ferritin proteins タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Complex produced by co-expression in E. coli |
|---|---|
| 由来(天然) | 生物種:  Haliangium ochraceum (バクテリア) Haliangium ochraceum (バクテリア) |
| 分子量 | 理論値: 1.8 MDa |
-分子 #1: Linocin_M18 bacteriocin protein
| 分子 | 名称: Linocin_M18 bacteriocin protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア)株: DSM 14365 / JCM 11303 / SMP-2 |
| 分子量 | 理論値: 28.844598 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDLLKRHLAP IVPDAWSAID EEAKEIFQGH LAGRKLVDFR GPFGWEYAAV NTGELRPIDD TPEDVDMKLR QVQPLAEVRV PFTLDVTEL DSVARGATNP DLDDVARAAE RMVEAEDSAI FHGWAQAGIK GIVDSTPHEA LAVASVSDFP RAVLSAADTL R KAGVTGPY ...文字列: MDLLKRHLAP IVPDAWSAID EEAKEIFQGH LAGRKLVDFR GPFGWEYAAV NTGELRPIDD TPEDVDMKLR QVQPLAEVRV PFTLDVTEL DSVARGATNP DLDDVARAAE RMVEAEDSAI FHGWAQAGIK GIVDSTPHEA LAVASVSDFP RAVLSAADTL R KAGVTGPY ALVLGPKAYD DLFAATQDGY PVAKQVQRLV VDGPLVRANA LAGALVMSMR GGDYELTVGQ DLSIGYAFHD RS KVELFVA ESFTFRVLEP GAAVHLRYA UniProtKB: Type 1 encapsulin shell protein |
-分子 #2: Haliangium ochraceum encapsulated ferritin
| 分子 | 名称: Haliangium ochraceum encapsulated ferritin / タイプ: protein_or_peptide / ID: 2 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア) Haliangium ochraceum (strain DSM 14365 / JCM 11303 / SMP-2) (バクテリア)株: DSM 14365 / JCM 11303 / SMP-2 |
| 分子量 | 理論値: 14.817368 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSEQLHEPA ELLSEETKNM HRALVTLIEE LEAVDWYQQR ADACSEPGLH DVLIHNKNEE VEHAMMTLEW IRRRSPVFDA HMRTYLFTE RPILELEEED TGSSSSVAAS PTSAPSHGSL GIGSLRQEGK ED UniProtKB: Encapsulated ferritin-like protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Solutions were prepared with MilliQ water and filtered using a 0.22 um filter. | |||||||||
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 詳細: grids were glow discharged for 30 seconds | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV 詳細: blot force -5, wait time 10 seconds and blot time of 3 seconds. | |||||||||
| 詳細 | Sample mono disperse as determined by SEC |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 11520 pixel / デジタル化 - サイズ - 縦: 8184 pixel / 撮影したグリッド数: 1 / 実像数: 8109 / 平均露光時間: 1.0 sec. / 平均電子線量: 40.509 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial fitting performed using chimera with real space refinement in Phenix. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 11.34 / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-7oeu: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)