+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of S.cerevisiae native alcohol dehydrogenase 1 (ADH1) in its tetrameric apo state | |||||||||
 マップデータ マップデータ | Local sharpen cryo-EM map of apo ADH1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ADH1 / tetramer / S.cerevisiae / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methylglyoxal reductase (NADH) / amino acid catabolic process to alcohol via Ehrlich pathway / octanol dehydrogenase (NAD+) activity / methylglyoxal reductase (NADH) activity / glycolytic fermentation to ethanol / butanol dehydrogenase (NAD+) activity / ethanol dehydrogenase (NAD+) activity / melatonin binding / alcohol dehydrogenase (NAD+) activity / allyl-alcohol dehydrogenase ...methylglyoxal reductase (NADH) / amino acid catabolic process to alcohol via Ehrlich pathway / octanol dehydrogenase (NAD+) activity / methylglyoxal reductase (NADH) activity / glycolytic fermentation to ethanol / butanol dehydrogenase (NAD+) activity / ethanol dehydrogenase (NAD+) activity / melatonin binding / alcohol dehydrogenase (NAD+) activity / allyl-alcohol dehydrogenase / allyl-alcohol dehydrogenase activity / alcohol dehydrogenase / zinc ion binding / identical protein binding / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
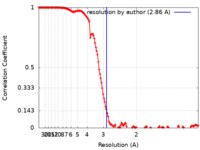
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.86 Å | |||||||||
 データ登録者 データ登録者 | Nzigou Mandouckou JA / Carroni M | |||||||||
| 資金援助 |  スウェーデン, 1件 スウェーデン, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of S.cerevisiae native alcohol dehydrogenase 1 (ADH1) in its tetrameric apo state 著者: Nzigou Mandouckou JA / Carroni M / Haeggstrom JZ / Thulasingam M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12591.map.gz emd_12591.map.gz | 214.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12591-v30.xml emd-12591-v30.xml emd-12591.xml emd-12591.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12591_fsc.xml emd_12591_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12591.png emd_12591.png | 78 KB | ||
| マスクデータ |  emd_12591_msk_1.map emd_12591_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12591.cif.gz emd-12591.cif.gz | 6.1 KB | ||
| その他 |  emd_12591_additional_1.map.gz emd_12591_additional_1.map.gz emd_12591_half_map_1.map.gz emd_12591_half_map_1.map.gz emd_12591_half_map_2.map.gz emd_12591_half_map_2.map.gz | 121.2 MB 226.2 MB 226.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12591 http://ftp.pdbj.org/pub/emdb/structures/EMD-12591 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12591 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12591 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12591_validation.pdf.gz emd_12591_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12591_full_validation.pdf.gz emd_12591_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_12591_validation.xml.gz emd_12591_validation.xml.gz | 22 KB | 表示 | |
| CIF形式データ |  emd_12591_validation.cif.gz emd_12591_validation.cif.gz | 28.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12591 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12591 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12591 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12591 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ntmMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12591.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12591.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local sharpen cryo-EM map of apo ADH1 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.6072 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_12591_msk_1.map emd_12591_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
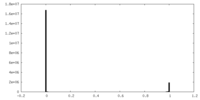
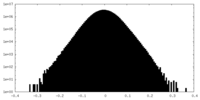
| 密度ヒストグラム |
-追加マップ: Unfiltered cryo-EM map of apo ADH1
| ファイル | emd_12591_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered cryo-EM map of apo ADH1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
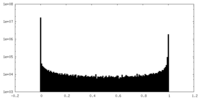
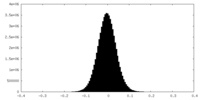
| 密度ヒストグラム |
-ハーフマップ: Half map A of cryo-EM map of apo ADH1
| ファイル | emd_12591_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of cryo-EM map of apo ADH1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
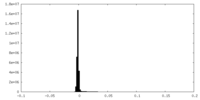
| 密度ヒストグラム |
-ハーフマップ: Half map B of cryo-EM map of apo ADH1
| ファイル | emd_12591_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of cryo-EM map of apo ADH1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
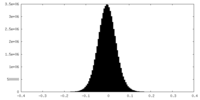
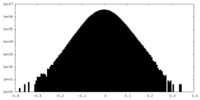
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tetrameric structure of native ADH1 from S. cereviciae in its apo...
| 全体 | 名称: Tetrameric structure of native ADH1 from S. cereviciae in its apo state. |
|---|---|
| 要素 |
|
-超分子 #1: Tetrameric structure of native ADH1 from S. cereviciae in its apo...
| 超分子 | 名称: Tetrameric structure of native ADH1 from S. cereviciae in its apo state. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Stable homotetramer, composed of four monomers labelled as A, B, C and D. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 147 KDa |
-分子 #1: Alcohol dehydrogenase 1
| 分子 | 名称: Alcohol dehydrogenase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: alcohol dehydrogenase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 36.759906 KDa |
| 配列 | 文字列: SIPETQKGVI FYESHGKLEY KDIPVPKPKA NELLINVKYS GVCHTDLHAW HGDWPLPVKL PLVGGHEGAG VVVGMGENVK GWKIGDYAG IKWLNGSCMA CEYCELGNES NCPHADLSGY THDGSFQQYA TADAVQAAHI PQGTDLAQVA PILCAGITVY K ALKSANLM ...文字列: SIPETQKGVI FYESHGKLEY KDIPVPKPKA NELLINVKYS GVCHTDLHAW HGDWPLPVKL PLVGGHEGAG VVVGMGENVK GWKIGDYAG IKWLNGSCMA CEYCELGNES NCPHADLSGY THDGSFQQYA TADAVQAAHI PQGTDLAQVA PILCAGITVY K ALKSANLM AGHWVAISGA AGGLGSLAVQ YAKAMGYRVL GIDGGEGKEE LFRSIGGEVF IDFTKEKDIV GAVLKATDGG AH GVINVSV SEAAIEASTR YVRANGTTVL VGMPAGAKCC SDVFNQVVKS ISIVGSYVGN RADTREALDF FARGLVKSPI KVV GLSTLP EIYEKMEKGQ IVGRYVVDTS K UniProtKB: Alcohol dehydrogenase 1 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 8 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Coma free - Residual tilt: 130.0 mrad |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 14964 / 平均露光時間: 2.5 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 1-347 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Model building and refinement were done using using CCP-EM software suite (Burnley et al, 2017). The X-ray structure of yeast ADH1 (PDB: 5ENV) was used as a starting model for model building. This model was docked into our Cryo-EM map using MolRep (Brown et al, 2014). The resulting starting model was manually adjusted in Coot (Emsley et al, 2010). REFMAC 5 (Brown et al, 2014) used for structure model refinement. |
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE / 温度因子: 123 |
| 得られたモデル |  PDB-7ntm: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)