+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12577 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ColicinE9 intact translocation complex | |||||||||
 マップデータ マップデータ | Output map from cryosparc local refinement job, sharpened using deepEMhancer tight target model | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | bacteriocin complex / import / membrane / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ABC-type vitamin B12 transporter activity / cell cycle / extrachromosomal circular DNA / colicin transmembrane transporter activity / cobalamin transport / transmembrane transporter complex / protein import / porin activity / pore complex / protein homotrimerization ...ABC-type vitamin B12 transporter activity / cell cycle / extrachromosomal circular DNA / colicin transmembrane transporter activity / cobalamin transport / transmembrane transporter complex / protein import / porin activity / pore complex / protein homotrimerization / monoatomic ion channel activity / monoatomic ion channel complex / lipopolysaccharide binding / cell outer membrane / disordered domain specific binding / protein transport / monoatomic ion transmembrane transport / endonuclease activity / killing of cells of another organism / 加水分解酵素; エステル加水分解酵素 / periplasmic space / defense response to bacterium / protein domain specific binding / cell division / lipid binding / calcium ion binding / protein-containing complex / metal ion binding / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |    | |||||||||
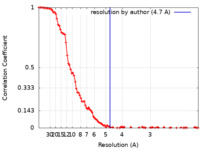
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Webby MN / Kleanthous C | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2021 ジャーナル: EMBO J / 年: 2021タイトル: Porin threading drives receptor disengagement and establishes active colicin transport through Escherichia coli OmpF. 著者: Marie-Louise R Francis / Melissa N Webby / Nicholas G Housden / Renata Kaminska / Emma Elliston / Boonyaporn Chinthammit / Natalya Lukoyanova / Colin Kleanthous /  要旨: Bacteria deploy weapons to kill their neighbours during competition for resources and to aid survival within microbiomes. Colicins were the first such antibacterial system identified, yet how these ...Bacteria deploy weapons to kill their neighbours during competition for resources and to aid survival within microbiomes. Colicins were the first such antibacterial system identified, yet how these bacteriocins cross the outer membrane (OM) of Escherichia coli is unknown. Here, by solving the structures of translocation intermediates via cryo-EM and by imaging toxin import, we uncover the mechanism by which the Tol-dependent nuclease colicin E9 (ColE9) crosses the bacterial OM. We show that threading of ColE9's disordered N-terminal domain through two pores of the trimeric porin OmpF causes the colicin to disengage from its primary receptor, BtuB, and reorganises the translocon either side of the membrane. Subsequent import of ColE9 through the lumen of a single OmpF subunit is driven by the proton-motive force, which is delivered by the TolQ-TolR-TolA-TolB assembly. Our study answers longstanding questions, such as why OmpF is a better translocator than OmpC, and reconciles the mechanisms by which both Tol- and Ton-dependent bacteriocins cross the bacterial outer membrane. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12577.map.gz emd_12577.map.gz | 74.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12577-v30.xml emd-12577-v30.xml emd-12577.xml emd-12577.xml | 24.7 KB 24.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12577_fsc.xml emd_12577_fsc.xml | 13 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12577.png emd_12577.png | 18.2 KB | ||
| マスクデータ |  emd_12577_msk_1.map emd_12577_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12577.cif.gz emd-12577.cif.gz | 7.6 KB | ||
| その他 |  emd_12577_additional_1.map.gz emd_12577_additional_1.map.gz emd_12577_half_map_1.map.gz emd_12577_half_map_1.map.gz emd_12577_half_map_2.map.gz emd_12577_half_map_2.map.gz | 41.6 MB 77.8 MB 77.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12577 http://ftp.pdbj.org/pub/emdb/structures/EMD-12577 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12577 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12577 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12577_validation.pdf.gz emd_12577_validation.pdf.gz | 958.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12577_full_validation.pdf.gz emd_12577_full_validation.pdf.gz | 958.4 KB | 表示 | |
| XML形式データ |  emd_12577_validation.xml.gz emd_12577_validation.xml.gz | 17.5 KB | 表示 | |
| CIF形式データ |  emd_12577_validation.cif.gz emd_12577_validation.cif.gz | 22.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12577 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12577 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12577 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12577 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12577.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12577.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Output map from cryosparc local refinement job, sharpened using deepEMhancer tight target model | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.047 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12577_msk_1.map emd_12577_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened output map from cryosparc local refinement job
| ファイル | emd_12577_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened output map from cryosparc local refinement job | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A from local refinement job in cryosparc
| ファイル | emd_12577_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A from local refinement job in cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B from local refinement job in cryosparc
| ファイル | emd_12577_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B from local refinement job in cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ColicinE9 intact translocon complex
| 全体 | 名称: ColicinE9 intact translocon complex |
|---|---|
| 要素 |
|
-超分子 #1: ColicinE9 intact translocon complex
| 超分子 | 名称: ColicinE9 intact translocon complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Bacteriocin colicinE9 bound to outer membrane protein receptor BtuB and translocator ompF. Disulphide linked to tolB. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 256 KDa |
-分子 #1: Outer membrane protein F
| 分子 | 名称: Outer membrane protein F / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 37.11425 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AEIYNKDGNK VDLYGKAVGL HYFSKGNGEN SYGGNGDMTY ARLGFKGETQ INSDLTGYGQ WEYNFQGNNS EGADAQTGNK TRLAFAGLK YADVGSFDYG RNYGVVYDAL GYTDMLPEFG GDTAYSDDFF VGRVGGVATY RNSNFFGLVD GLNFAVQYLG K NERDTARR ...文字列: AEIYNKDGNK VDLYGKAVGL HYFSKGNGEN SYGGNGDMTY ARLGFKGETQ INSDLTGYGQ WEYNFQGNNS EGADAQTGNK TRLAFAGLK YADVGSFDYG RNYGVVYDAL GYTDMLPEFG GDTAYSDDFF VGRVGGVATY RNSNFFGLVD GLNFAVQYLG K NERDTARR SNGDGVGGSI SYEYEGFGIV GAYGAADRTN LQEAQPLGNG KKAEQWATGL KYDANNIYLA ANYGETRNAT PI TNKFTNT SGFANKTQDV LLVAQYQFDF GLRPSIAYTK SKAKDVEGIG DVDLVNYFEV GATYYFNKNM STYVDYIINQ IDS DNKLGV GSDDTVAVGI VYQF UniProtKB: Outer membrane porin F |
-分子 #2: Colicin-E9
| 分子 | 名称: Colicin-E9 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.707246 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGGDGRGHN TGAHSTSGNI NGGPTGIGVS GGCSDGSGWS SENNPWGGGS GSGIHWGGGS GRGNGGGNGN SGGGSGTGGN LSAVAAPVA FGFPALSTPG AGGLAVSISA SELSAAIAGI IAKLKKVNLK FTPFGVVLSS LIPSEIAKDD PNMMSKIVTS L PADDITES ...文字列: MSGGDGRGHN TGAHSTSGNI NGGPTGIGVS GGCSDGSGWS SENNPWGGGS GSGIHWGGGS GRGNGGGNGN SGGGSGTGGN LSAVAAPVA FGFPALSTPG AGGLAVSISA SELSAAIAGI IAKLKKVNLK FTPFGVVLSS LIPSEIAKDD PNMMSKIVTS L PADDITES PVSSLPLDKA TVNVNVRVVD DVKDERQNIS VVSGVPMSVP VVDAKPTERP GVFTASIPGA PVLNISVNDS TP AVQTLSP GVTNNTDKDV RPAGFTQGGN TRDAVIRFPK DSGHNAVYVS VSDVLSPDQV KQRQDEENRR QQEWDATHPV EAA ERNYER ARAELNQANE DVARNQERQA KAVQVYNSRK SELDAANKTL ADAIAEIKQF NRFAHDPMAG GHRMWQMAGL KAQR AQTDV NNKQAAFDAA AKEKSDADAA LSAAQERRKQ KENKEKDAKD KLDKESKRNK PGKATGKGKP VGDKWLDDAG KDSGA PIPD RIADKLRDKE FKSFDDFRKA VWEEVSKDPE LSKNLNPSNK SSVSKGYSPF TPKNQQVGGR KVYELHHDKP ISQGGE VYD MDNIRVTTPK RHIDIHRGK UniProtKB: Colicin-E9 |
-分子 #3: Tol-Pal system protein TolB
| 分子 | 名称: Tol-Pal system protein TolB / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46.000285 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKQALRVAFG FLILWASVLH AEVRIVIDSG VDSGRPIGVV PFQWAGPGAA PEDIGGIVAA DLRNSGKFNP LDRARLPQQP GSAQEVQPA AWSALGIDAV VVGQVTPNPD GSYNVAYQLV DTGGAPGTVL AQNSYKVNKQ WLRYAGHTAS DEVFEKLTGI K GAFRTRIA ...文字列: MKQALRVAFG FLILWASVLH AEVRIVIDSG VDSGRPIGVV PFQWAGPGAA PEDIGGIVAA DLRNSGKFNP LDRARLPQQP GSAQEVQPA AWSALGIDAV VVGQVTPNPD GSYNVAYQLV DTGGAPGTVL AQNSYKVNKQ WLRYAGHTAS DEVFEKLTGI K GAFRTRIA YVVQTNGGQF PYELRVSDYD GYNQFVVHRS PQCLMSPAWS PDGSKLAYVT FESGRSALVI QTLANGAVRQ VA SFPRHNG APAFSPDGSK LAFALSKTGS LNLYVMDLAS GQIRQVTDGR SNNTEPTWFP DSQNLAFTSD QAGRPQVYKV NIN GGAPQR ITWEGSQNQD ADVSSDGKFM VMVSSNGGQQ HIAKQDLATG GVQVLSSTFL DETPSLAPNG TMVIYSSSQG MGSV LNLVS TDGRFKARLP ATDGQVKFPA WSPYL UniProtKB: Tol-Pal system protein TolB |
-分子 #4: Vitamin B12 transporter BtuB
| 分子 | 名称: Vitamin B12 transporter BtuB / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 66.38618 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QDTSPDTLVV TANRFEQPRS TVLAPTTVVT RQDIDRWQST SVNDVLRRLP GVDITQNGGS GQLSSIFIRG TNASHVLVLI DGVRLNLAG VSGSADLSQF PIALVQRVEY IRGPRSAVYG SDAIGGVVNI ITTRDEPGTE ISAGWGSNSY QNYDVSTQQQ L GDKTRVTL ...文字列: QDTSPDTLVV TANRFEQPRS TVLAPTTVVT RQDIDRWQST SVNDVLRRLP GVDITQNGGS GQLSSIFIRG TNASHVLVLI DGVRLNLAG VSGSADLSQF PIALVQRVEY IRGPRSAVYG SDAIGGVVNI ITTRDEPGTE ISAGWGSNSY QNYDVSTQQQ L GDKTRVTL LGDYAHTHGY DVVAYGNTGT QAQTDNDGFL SKTLYGALEH NFTDAWSGFV RGYGYDNRTN YDAYYSPGSP LL DTRKLYS QSWDAGLRYN GELIKSQLIT SYSHSKDYNY DPHYGRYDSS ATLDEMKQYT VQWANNVIVG HGSIGAGVDW QKQ TTTPGT GYVEDGYDQR NTGIYLTGLQ QVGDFTFEGA ARSDDNSQFG RHGTWQTSAG WEFIEGYRFI ASYGTSYKAP NLGQ LYGFY GNPNLDPEKS KQWEGAFEGL TAGVNWRISG YRNDVSDLID YDDHTLKYYN EGKARIKGVE ATANFDTGPL THTVS YDYV DARNAITDTP LLRRAKQQVK YQLDWQLYDF DWGITYQYLG TRYDKDYSSY PYQTVKMGGV SLWDLAVAYP VTSHLT VRG KIANLFDKDY ETVYGYQTAG REYTLSGSYT F UniProtKB: Vitamin B12 transporter BtuB |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: GRAPHENE OXIDE / 支持フィルム - #0 - トポロジー: CONTINUOUS / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: CARBON / 支持フィルム - #1 - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: PELCO easiGlow, Ted Pella Inc, USA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 92 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: 3 uL of diluted translocon preparation were applied on freshly glow discharged grids coated with graphene oxide (as described in https://doi.org/10.1038/s41594-019-0355-2); after 30sec ...詳細: 3 uL of diluted translocon preparation were applied on freshly glow discharged grids coated with graphene oxide (as described in https://doi.org/10.1038/s41594-019-0355-2); after 30sec waiting grids were blotted for 8-10 using -10 force and plunge frozen in liquid ethane. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 2 / 平均露光時間: 10.0 sec. / 平均電子線量: 49.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm 最大 デフォーカス(補正後): 3.8000000000000003 µm 最小 デフォーカス(補正後): 1.23 µm / 倍率(補正後): 47755 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 47619 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)