[English] 日本語
 Yorodumi
Yorodumi- EMDB-12348: 1918 H1N1 Viral influenza polymerase heterotrimer - Replicase (cl... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-12348 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | 1918 H1N1 Viral influenza polymerase heterotrimer - Replicase (class 3) | ||||||||||||
 Map data Map data | Original unsharpened map from cryoSPARC | ||||||||||||
 Sample Sample |
| ||||||||||||
 Keywords Keywords | Influenza / RNA polymerase / H1N1 / 1918 / VIRAL PROTEIN | ||||||||||||
| Function / homology |  Function and homology information Function and homology informationIgG binding / cap snatching / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / virion component / endonuclease activity / Hydrolases; Acting on ester bonds ...IgG binding / cap snatching / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / virion component / endonuclease activity / Hydrolases; Acting on ester bonds / host cell cytoplasm / symbiont-mediated suppression of host gene expression / viral translational frameshifting / hydrolase activity / RNA-directed RNA polymerase / viral RNA genome replication / nucleotide binding / RNA-directed RNA polymerase activity / DNA-templated transcription / host cell nucleus / RNA binding / extracellular region / metal ion binding Similarity search - Function | ||||||||||||
| Biological species |  Influenza A virus (strain A/Brevig Mission/1/1918 H1N1) / Influenza A virus (strain A/Brevig Mission/1/1918 H1N1) /   Influenza A virus (A/Brevig Mission/1/1918(H1N1)) Influenza A virus (A/Brevig Mission/1/1918(H1N1)) | ||||||||||||
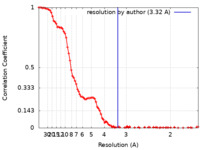
| Method | single particle reconstruction / cryo EM / Resolution: 3.32 Å | ||||||||||||
 Authors Authors | Keown JR / Carrique L | ||||||||||||
| Funding support |  United Kingdom, 3 items United Kingdom, 3 items
| ||||||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2022 Journal: Nat Commun / Year: 2022Title: Mapping inhibitory sites on the RNA polymerase of the 1918 pandemic influenza virus using nanobodies. Authors: Jeremy R Keown / Zihan Zhu / Loïc Carrique / Haitian Fan / Alexander P Walker / Itziar Serna Martin / Els Pardon / Jan Steyaert / Ervin Fodor / Jonathan M Grimes /    Abstract: Influenza A viruses cause seasonal epidemics and global pandemics, representing a considerable burden to healthcare systems. Central to the replication cycle of influenza viruses is the viral RNA- ...Influenza A viruses cause seasonal epidemics and global pandemics, representing a considerable burden to healthcare systems. Central to the replication cycle of influenza viruses is the viral RNA-dependent RNA polymerase which transcribes and replicates the viral RNA genome. The polymerase undergoes conformational rearrangements and interacts with viral and host proteins to perform these functions. Here we determine the structure of the 1918 influenza virus polymerase in transcriptase and replicase conformations using cryo-electron microscopy (cryo-EM). We then structurally and functionally characterise the binding of single-domain nanobodies to the polymerase of the 1918 pandemic influenza virus. Combining these functional and structural data we identify five sites on the polymerase which are sensitive to inhibition by nanobodies. We propose that the binding of nanobodies at these sites either prevents the polymerase from assuming particular functional conformations or interactions with viral or host factors. The polymerase is highly conserved across the influenza A subtypes, suggesting these sites as effective targets for potential influenza antiviral development. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_12348.map.gz emd_12348.map.gz | 32 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-12348-v30.xml emd-12348-v30.xml emd-12348.xml emd-12348.xml | 24.3 KB 24.3 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_12348_fsc.xml emd_12348_fsc.xml | 11.8 KB | Display |  FSC data file FSC data file |
| Images |  emd_12348.png emd_12348.png | 94.2 KB | ||
| Filedesc metadata |  emd-12348.cif.gz emd-12348.cif.gz | 8 KB | ||
| Others |  emd_12348_additional_1.map.gz emd_12348_additional_1.map.gz | 59.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12348 http://ftp.pdbj.org/pub/emdb/structures/EMD-12348 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12348 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12348 | HTTPS FTP |
-Related structure data
| Related structure data |  7ni0MC  7nfqC  7nfrC  7nftC  7nhaC  7nhcC  7nhxC  7nikC  7nilC  7nirC  7nisC  7nj3C  7nj4C  7nj5C  7nj7C  7nk1C  7nk2C  7nk4C  7nk6C  7nk8C  7nkaC  7nkcC  7nkiC  7nkrC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_12348.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_12348.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Original unsharpened map from cryoSPARC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.825 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Additional map: Map generated by deepEMhancer using the wideTarget function.
| File | emd_12348_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Map generated by deepEMhancer using the wideTarget function. | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
+Entire : 1918 Influenza virus polymerase heterotirmer in complex with vRNA...
+Supramolecule #1: 1918 Influenza virus polymerase heterotirmer in complex with vRNA...
+Supramolecule #2: Polymerase acidic protein
+Supramolecule #3: RNA-directed RNA polymerase catalytic subunit
+Supramolecule #4: Polymerase basic protein 2
+Supramolecule #5: RNA
+Macromolecule #1: Polymerase acidic protein
+Macromolecule #2: RNA-directed RNA polymerase catalytic subunit
+Macromolecule #3: Polymerase basic protein 2,Immunoglobulin G-binding protein A
+Macromolecule #4: RNA (5'-R(P*GP*GP*CP*CP*UP*GP*CP*U)-3')
+Macromolecule #5: RNA (5'-R(P*AP*GP*UP*AP*GP*AP*AP*AP*CP*AP*AP*GP*GP*CP*C)-3')
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.3 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.5 Component:
| |||||||||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 298 K / Instrument: FEI VITROBOT MARK IV / Details: 3.5 second blot and 3 microlitres sample.. |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Detector mode: COUNTING / Digitization - Dimensions - Width: 3838 pixel / Digitization - Dimensions - Height: 3710 pixel / Number grids imaged: 1 / Number real images: 9413 / Average exposure time: 5.0 sec. / Average electron dose: 61.5 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal magnification: 165000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller
































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)