+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12255 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | pT=4, Q=10 quasi-symmetric bacterial microcompartment particle | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Kalnins G / Cesle EE | |||||||||
| 資金援助 |  Latvia, Latvia,  チェコ, 2件 チェコ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Protein Sci / 年: 2021 ジャーナル: Protein Sci / 年: 2021タイトル: Variety of size and form of GRM2 bacterial microcompartment particles. 著者: Eva Emilija Cesle / Anatolij Filimonenko / Kaspars Tars / Gints Kalnins /   要旨: Bacterial microcompartments (BMCs) are bacterial organelles involved in enzymatic processes, such as carbon fixation, choline, ethanolamine and propanediol degradation, and others. Formed of a semi- ...Bacterial microcompartments (BMCs) are bacterial organelles involved in enzymatic processes, such as carbon fixation, choline, ethanolamine and propanediol degradation, and others. Formed of a semi-permeable protein shell and an enzymatic core, they can enhance enzyme performance and protect the cell from harmful intermediates. With the ability to encapsulate non-native enzymes, BMCs show high potential for applied use. For this goal, a detailed look into shell form variability is significant to predict shell adaptability. Here we present four novel 3D cryo-EM maps of recombinant Klebsiella pneumoniae GRM2 BMC shell particles with the resolution in range of 9 to 22 Å and nine novel 2D classes corresponding to discrete BMC shell forms. These structures reveal icosahedral, elongated, oblate, multi-layered and polyhedral traits of BMCs, indicating considerable variation in size and form as well as adaptability during shell formation processes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12255.map.gz emd_12255.map.gz | 80.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12255-v30.xml emd-12255-v30.xml emd-12255.xml emd-12255.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
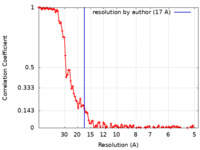
| FSC (解像度算出) |  emd_12255_fsc.xml emd_12255_fsc.xml | 13.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12255.png emd_12255.png | 110.9 KB | ||
| その他 |  emd_12255_half_map_1.map.gz emd_12255_half_map_1.map.gz emd_12255_half_map_2.map.gz emd_12255_half_map_2.map.gz | 81 MB 80.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12255 http://ftp.pdbj.org/pub/emdb/structures/EMD-12255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12255 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12255_validation.pdf.gz emd_12255_validation.pdf.gz | 388.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12255_full_validation.pdf.gz emd_12255_full_validation.pdf.gz | 388 KB | 表示 | |
| XML形式データ |  emd_12255_validation.xml.gz emd_12255_validation.xml.gz | 16.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12255 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12255 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12255.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12255.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.44 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_12255_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
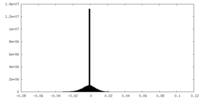
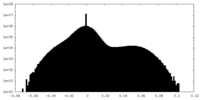
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12255_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
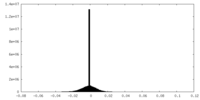
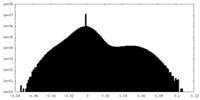
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : pT=4, Q=10 quasi-symmetric bacterial microcompartment particles
| 全体 | 名称: pT=4, Q=10 quasi-symmetric bacterial microcompartment particles |
|---|---|
| 要素 |
|
-超分子 #1: pT=4, Q=10 quasi-symmetric bacterial microcompartment particles
| 超分子 | 名称: pT=4, Q=10 quasi-symmetric bacterial microcompartment particles タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: bacterial microcompartment particle obtained by recombinant expression of pentameric EutN and hexameric cmcC subunits |
|---|---|
| 由来(天然) | 生物種:  Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) |
| 組換発現 | 生物種:  |
-分子 #1: cmcC/PduA/ccmK BMC-H bacterial microcompartment protein
| 分子 | 名称: cmcC/PduA/ccmK BMC-H bacterial microcompartment protein タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKEALGLIET KGLVACIEAA DAMCKAANVE LIGYENVGSG LVTAMVKGDV GAVNAAVDSG VEAAKRIGKV VSSRVIARPH NDIEKIAGST KHKSLRPHNA |
-分子 #2: cmcD/EutN BMC-P bacterial microcompartment protein
| 分子 | 名称: cmcD/EutN BMC-P bacterial microcompartment protein / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MILAKVTGHV VATQKCDELR GSNLLLITRL DDKQQPMKDQ TWVAVDNVGA GMHDIVLAEE YFALNKDRYK AMSVVAIVEK VFRDTEQE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 4s before plunging. | ||||||
| 詳細 | Sample was purified with ultracentrifugation and gel filtration |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 実像数: 3240 / 平均露光時間: 1.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm 最小 デフォーカス(補正後): 0.7000000000000001 µm 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 120000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)