+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1223 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The dynamics of signal triggering in a gp130-receptor complex. | |||||||||
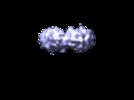
 マップデータ マップデータ | This is a cEM map of the extracellular portion of the IL11 hexameric complex | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 30.0 Å | |||||||||
 データ登録者 データ登録者 | Matadeen R / Hon W / Jones EY / Fuller S | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2007 ジャーナル: Structure / 年: 2007タイトル: The dynamics of signal triggering in a gp130-receptor complex. 著者: Rishi Matadeen / Wai-Ching Hon / John K Heath / E Yvonne Jones / Stephen Fuller /  要旨: gp130 is a shared signal-transducing membrane-associated receptor for several hematopoietic cytokines. The 30 A resolution cryo-electron microscopy (cryo-EM) structure of the Interleukin 11(IL-11)-IL- ...gp130 is a shared signal-transducing membrane-associated receptor for several hematopoietic cytokines. The 30 A resolution cryo-electron microscopy (cryo-EM) structure of the Interleukin 11(IL-11)-IL-11 Receptor-gp130 extracellular complex reveals the architecture and dynamics of this gp130-containing signaling complex. Normal-mode analysis reveals a repertoire of conformational changes that could function in signal triggering. This suggests a concerted mechanism of signaling involving all the components of the complex. This could provide a general mechanism of signal transfer for cytokines utilizing the JAK-STAT signaling cascade. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1223.map.gz emd_1223.map.gz | 499.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1223-v30.xml emd-1223-v30.xml emd-1223.xml emd-1223.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1223.gif 1223.gif | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1223 http://ftp.pdbj.org/pub/emdb/structures/EMD-1223 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1223 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1223 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1223_validation.pdf.gz emd_1223_validation.pdf.gz | 209.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1223_full_validation.pdf.gz emd_1223_full_validation.pdf.gz | 208.7 KB | 表示 | |
| XML形式データ |  emd_1223_validation.xml.gz emd_1223_validation.xml.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1223 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1223 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1223 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1223 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1223.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1223.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a cEM map of the extracellular portion of the IL11 hexameric complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : IL11-IL11-R-gp130
| 全体 | 名称: IL11-IL11-R-gp130 |
|---|---|
| 要素 |
|
-超分子 #1000: IL11-IL11-R-gp130
| 超分子 | 名称: IL11-IL11-R-gp130 / タイプ: sample / ID: 1000 / 集合状態: hexamer / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 270 KDa |
-分子 #1: Interleukin-11
| 分子 | 名称: Interleukin-11 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: IL-11 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #2: Interleukin-11 Receptor
| 分子 | 名称: Interleukin-11 Receptor / タイプ: protein_or_peptide / ID: 2 / Name.synonym: IL-11R / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #3: Glycoprotein 130
| 分子 | 名称: Glycoprotein 130 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: gp130 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 5mM HEPES 100mM NaCl 1mM DTT 0.25% B-octylglucopyranoside |
| グリッド | 詳細: 300 mesh holy carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 100,000 times magnification |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - サンプリング間隔: 8.3 µm / 実像数: 15 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50100 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 3.0 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: side entry / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 30.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Imagic / 使用した粒子像数: 830 |
| 最終 2次元分類 | クラス数: 100 |
-原子モデル構築 1
| ソフトウェア | 名称: URO |
|---|---|
| 詳細 | Protocol: Rigid Body |
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















