+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12061 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast 80S ribosome bound to eEF3 and A/A- and P/E-tRNAs (PRE-2). | ||||||||||||||||||
 マップデータ マップデータ | Cryo-EM structure of the S. cerevisiae 80S ribosome in complex with eEF3 and A/A- and P/E-site tRNAs (postprocessed masked) | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
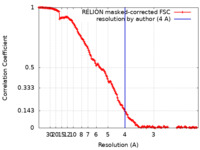
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Ranjan N / Pochopien AA / Wu CC / Beckert B / Blanchet S / Green R / Rodnina MV / Wilson DN | ||||||||||||||||||
| 資金援助 | 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2021 ジャーナル: EMBO J / 年: 2021タイトル: Yeast translation elongation factor eEF3 promotes late stages of tRNA translocation. 著者: Namit Ranjan / Agnieszka A Pochopien / Colin Chih-Chien Wu / Bertrand Beckert / Sandra Blanchet / Rachel Green / Marina V Rodnina / Daniel N Wilson /   要旨: In addition to the conserved translation elongation factors eEF1A and eEF2, fungi require a third essential elongation factor, eEF3. While eEF3 has been implicated in tRNA binding and release at the ...In addition to the conserved translation elongation factors eEF1A and eEF2, fungi require a third essential elongation factor, eEF3. While eEF3 has been implicated in tRNA binding and release at the ribosomal A and E sites, its exact mechanism of action is unclear. Here, we show that eEF3 acts at the mRNA-tRNA translocation step by promoting the dissociation of the tRNA from the E site, but independent of aminoacyl-tRNA recruitment to the A site. Depletion of eEF3 in vivo leads to a general slowdown in translation elongation due to accumulation of ribosomes with an occupied A site. Cryo-EM analysis of native eEF3-ribosome complexes shows that eEF3 facilitates late steps of translocation by favoring non-rotated ribosomal states, as well as by opening the L1 stalk to release the E-site tRNA. Additionally, our analysis provides structural insights into novel translation elongation states, enabling presentation of a revised yeast translation elongation cycle. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
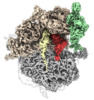
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12061.map.gz emd_12061.map.gz | 32.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12061-v30.xml emd-12061-v30.xml emd-12061.xml emd-12061.xml | 15.4 KB 15.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12061_fsc.xml emd_12061_fsc.xml | 14.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12061.png emd_12061.png | 211.9 KB | ||
| その他 |  emd_12061_additional_1.map.gz emd_12061_additional_1.map.gz emd_12061_half_map_1.map.gz emd_12061_half_map_1.map.gz emd_12061_half_map_2.map.gz emd_12061_half_map_2.map.gz | 224.8 MB 225.5 MB 225.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12061 http://ftp.pdbj.org/pub/emdb/structures/EMD-12061 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12061 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12061 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12061_validation.pdf.gz emd_12061_validation.pdf.gz | 435.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12061_full_validation.pdf.gz emd_12061_full_validation.pdf.gz | 435.1 KB | 表示 | |
| XML形式データ |  emd_12061_validation.xml.gz emd_12061_validation.xml.gz | 23.1 KB | 表示 | |
| CIF形式データ |  emd_12061_validation.cif.gz emd_12061_validation.cif.gz | 29.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12061 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12061 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12061 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12061 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12061.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12061.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the S. cerevisiae 80S ribosome in complex with eEF3 and A/A- and P/E-site tRNAs (postprocessed masked) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.084 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Cryo-EM structure of the S. cerevisiae 80S ribosome...
| ファイル | emd_12061_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the S. cerevisiae 80S ribosome in complex with eEF3 and A/A- and P/E-site tRNAs (refinement) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM structure of the S. cerevisiae 80S ribosome...
| ファイル | emd_12061_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the S. cerevisiae 80S ribosome in complex with eEF3 and A/A- and P/E-site tRNAs (half map 1) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM structure of the S. cerevisiae 80S ribosome...
| ファイル | emd_12061_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the S. cerevisiae 80S ribosome in complex with eEF3 and A/A- and P/E-site tRNAs (half map 2) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the S. cerevisiae 80S ribosome in complex wi...
| 全体 | 名称: Cryo-EM structure of the S. cerevisiae 80S ribosome in complex with eEF3 and A/A- and P/P-site tRNAs. |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the S. cerevisiae 80S ribosome in complex wi...
| 超分子 | 名称: Cryo-EM structure of the S. cerevisiae 80S ribosome in complex with eEF3 and A/A- and P/P-site tRNAs. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)