+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11996 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | OsBOR3 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Bicarbonate transporter, eukaryotic / Bicarbonate transporter-like, transmembrane domain / HCO3- transporter integral membrane domain / solute:inorganic anion antiporter activity / membrane => GO:0016020 / Boron transporter 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
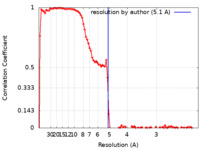
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.1 Å | |||||||||
 データ登録者 データ登録者 | Byrne B / Barritt JD | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2021 ジャーナル: Sci Rep / 年: 2021タイトル: Structural and functional insights into the mechanism of action of plant borate transporters. 著者: Savvas Saouros / Thotegowdanapalya C Mohan / Cristina Cecchetti / Silke Lehmann / Joseph D Barrit / Nicola J Scull / Paul Simpson / Yilmaz Alguel / Alexander D Cameron / Alexandra M E Jones / Bernadette Byrne /   要旨: Boron has essential roles in plant growth and development. BOR proteins are key in the active uptake and distribution of boron, and regulation of intracellular boron concentrations. However, their ...Boron has essential roles in plant growth and development. BOR proteins are key in the active uptake and distribution of boron, and regulation of intracellular boron concentrations. However, their mechanism of action remains poorly studied. BOR proteins are homologues of the human SLC4 family of transporters, which includes well studied mammalian transporters such as the human Anion Exchanger 1 (hAE1). Here we generated Arabidopsis thaliana BOR1 (AtBOR1) variants based (i) on known disease causing mutations of hAE1 (S466R, A500R) and (ii) a loss of function mutation (D311A) identified in the yeast BOR protein, ScBOR1p. The AtBOR1 variants express in yeast and localise to the plasma membrane, although both S466R and A500R exhibit lower expression than the WT AtBOR1 and D311A. The D311A, S466R and A500R mutations result in a loss of borate efflux activity in a yeast bor1p knockout strain. A. thaliana plants containing these three individual mutations exhibit substantially decreased growth phenotypes in soil under conditions of low boron. These data confirm an important role for D311 in the function of the protein and show that mutations equivalent to disease-causing mutations in hAE1 have major effects in AtBOR1. We also obtained a low resolution cryo-EM structure of a BOR protein from Oryza sativa, OsBOR3, lacking the 30 C-terminal amino acid residues. This structure confirms the gate and core domain organisation previously observed for related proteins, and is strongly suggestive of an inward facing conformation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11996.map.gz emd_11996.map.gz | 3.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11996-v30.xml emd-11996-v30.xml emd-11996.xml emd-11996.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11996_fsc.xml emd_11996_fsc.xml | 11.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11996.png emd_11996.png | 100.5 KB | ||
| マスクデータ |  emd_11996_msk_1.map emd_11996_msk_1.map | 59.6 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11996 http://ftp.pdbj.org/pub/emdb/structures/EMD-11996 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11996 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11996 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11996_validation.pdf.gz emd_11996_validation.pdf.gz | 247.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11996_full_validation.pdf.gz emd_11996_full_validation.pdf.gz | 246.2 KB | 表示 | |
| XML形式データ |  emd_11996_validation.xml.gz emd_11996_validation.xml.gz | 10.6 KB | 表示 | |
| CIF形式データ |  emd_11996_validation.cif.gz emd_11996_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11996 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11996 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11996 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11996 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11996.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11996.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11996_msk_1.map emd_11996_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Borate transporter 3 from Oryza sativa (osBOR3)
| 全体 | 名称: Borate transporter 3 from Oryza sativa (osBOR3) |
|---|---|
| 要素 |
|
-超分子 #1: Borate transporter 3 from Oryza sativa (osBOR3)
| 超分子 | 名称: Borate transporter 3 from Oryza sativa (osBOR3) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Homodimer of the osBOR3 integral membrane protein in LMNG detergent belt. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #1: Oryza sativa borate transporter 3
| 分子 | 名称: Oryza sativa borate transporter 3 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEESFVPLRG IKNDLHGRL Q CYKQDWTG GF RAGIRIL APT TYIFFA SAIP VISFG EQLER NTDG VLTAVQ TLA STALCGI IH SFLGGQPL L ILGVAEPTV LMYTFMFNFA KDRPDLGRR L FLAWTGWV CV WTAILLF LLA ILGACS IINR FTRIA ...文字列: MEESFVPLRG IKNDLHGRL Q CYKQDWTG GF RAGIRIL APT TYIFFA SAIP VISFG EQLER NTDG VLTAVQ TLA STALCGI IH SFLGGQPL L ILGVAEPTV LMYTFMFNFA KDRPDLGRR L FLAWTGWV CV WTAILLF LLA ILGACS IINR FTRIA GELFG LLIA MLFMQQ AIK GLVDEFR IP ERENRKAL E FVSSWRFAN GMFAIVLSFG LLLTALRSR K ARSWRYGT GW LRGFIAD YGV PLMVLV WTGV SYIPY GSVPK GIPR RLFSPN PWS PGAYDNW TV IRDMPNVP L LYIIGAFIP ATMIAVLYYF DHSVASQLA Q QKEFNLRK PP SFHYDLL LLG FLTLLC GLIG IPPAN GVIPQ SPMH TKSLAT LKH QLLRNRL VA TARQSMSQ N ASLSQLYGS MQEAYQQMQT PLIYQQPSV K GLNELKDS TV QMASSMG NID APVDET VFDI EKEID DLLPI EVKE QRLSNL LQA SMVGGCV AA MPLLKKIP T SVLWGYFAF MAIESLPGNQ FWERILLLF T APSRRYKV LE EYHTTFV ETV PFKTIA MFTL FQTMY LLVCF GITW IPIAGV LFP LMIMLLV PV RQYILPKL F KGAHLTDLD AAEYEESPAI PFIAAQDID V ALARTQSA EI LDDIVTR SRG EI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 20.265 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 3 second blot time -2 blot force. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 6285 / 平均露光時間: 4.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X