+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11971 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
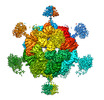
| タイトル | Stressosome complex from Listeria innocua | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Stressosome / stress sensing / general stress response / phosphorylation cascade / SIGNALING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Listeria innocua Clip11262 (バクテリア) / Listeria innocua Clip11262 (バクテリア) /  Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア) Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.86 Å | |||||||||
 データ登録者 データ登録者 | Miksys A / Fu L | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2022 ジャーナル: Commun Biol / 年: 2022タイトル: Molecular insights into intra-complex signal transmission during stressosome activation. 著者: Algirdas Miksys / Lifei Fu / M Gregor Madej / Duarte N Guerreiro / Susann Kaltwasser / Maria Conway / Sema Ejder / Astrid Bruckmann / Jon Marles-Wright / Richard J Lewis / Conor O'Byrne / Jan ...著者: Algirdas Miksys / Lifei Fu / M Gregor Madej / Duarte N Guerreiro / Susann Kaltwasser / Maria Conway / Sema Ejder / Astrid Bruckmann / Jon Marles-Wright / Richard J Lewis / Conor O'Byrne / Jan Pané-Farré / Christine Ziegler /    要旨: The stressosome is a pseudo-icosahedral megadalton bacterial stress-sensing protein complex consisting of several copies of two STAS-domain proteins, RsbR and RsbS, and the kinase RsbT. Upon ...The stressosome is a pseudo-icosahedral megadalton bacterial stress-sensing protein complex consisting of several copies of two STAS-domain proteins, RsbR and RsbS, and the kinase RsbT. Upon perception of environmental stress multiple copies of RsbT are released from the surface of the stressosome. Free RsbT activates downstream proteins to elicit a global cellular response, such as the activation of the general stress response in Gram-positive bacteria. The molecular events triggering RsbT release from the stressosome surface remain poorly understood. Here we present the map of Listeria innocua RsbR1/RsbS complex at resolutions of 3.45 Å for the STAS domain core in icosahedral symmetry and of 3.87 Å for the STAS domain and N-terminal sensors in D2 symmetry, respectively. The structure reveals a conformational change in the STAS domain linked to phosphorylation in RsbR. Docking studies indicate that allosteric RsbT binding to the conformationally flexible N-terminal sensor domain of RsbR affects the affinity of RsbS towards RsbT. Our results bring to focus the molecular events within the stressosome complex and further our understanding of this ubiquitous signaling hub. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11971.map.gz emd_11971.map.gz | 200.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11971-v30.xml emd-11971-v30.xml emd-11971.xml emd-11971.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11971.png emd_11971.png | 167.5 KB | ||
| Filedesc metadata |  emd-11971.cif.gz emd-11971.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11971 http://ftp.pdbj.org/pub/emdb/structures/EMD-11971 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11971 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11971 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11971_validation.pdf.gz emd_11971_validation.pdf.gz | 587.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11971_full_validation.pdf.gz emd_11971_full_validation.pdf.gz | 587.5 KB | 表示 | |
| XML形式データ |  emd_11971_validation.xml.gz emd_11971_validation.xml.gz | 7.3 KB | 表示 | |
| CIF形式データ |  emd_11971_validation.cif.gz emd_11971_validation.cif.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11971 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11971 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11971 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11971 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11971.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11971.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0635 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : stressosome complex RsbR and RsbS
| 全体 | 名称: stressosome complex RsbR and RsbS |
|---|---|
| 要素 |
|
-超分子 #1: stressosome complex RsbR and RsbS
| 超分子 | 名称: stressosome complex RsbR and RsbS / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Listeria innocua Clip11262 (バクテリア) Listeria innocua Clip11262 (バクテリア) |
| 分子量 | 理論値: 1.5 MDa |
-分子 #1: RsbR protein
| 分子 | 名称: RsbR protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア) Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア)株: ATCC BAA-680 / CLIP 11262 |
| 分子量 | 理論値: 31.704586 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP ...文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP LIGTIDTERA KLIIENLLIG VVKNRSEVVL IDITGVPVVD TMVAHHIIQA SEAVRLVGCQ AMLVGIRPEI AQ TIVNLGI ELDQIITTNT MKKGMERALA LTNREIVEKE G UniProtKB: RsbR protein |
-分子 #2: RsbR protein
| 分子 | 名称: RsbR protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア) Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア)株: ATCC BAA-680 / CLIP 11262 |
| 分子量 | 理論値: 31.784566 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP ...文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP LIGTIDTERA KLIIENLLIG VVKNRSEVVL IDITGVPVVD TMVAHHIIQA SEAVRLVGCQ AMLVGIRPEI AQ (TPO)IVNLGI ELDQIITTNT MKKGMERALA LTNREIVEKE G UniProtKB: RsbR protein |
-分子 #3: RsbS protein
| 分子 | 名称: RsbS protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 20 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア) Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア)株: ATCC BAA-680 / CLIP 11262 |
| 分子量 | 理論値: 12.606707 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGIPILKLGE CLLISIQSEL DDHTAVEFQE DLLAKIHETS ARGVVIDITS IDFIDSFIAK ILGDVVSMSK LMGAKVVVTG IQPAVAITL IELGITFSGV LSAMDLESGL EKLKQELGE UniProtKB: RsbS protein |
-分子 #4: RsbR protein
| 分子 | 名称: RsbR protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア) Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア)株: ATCC BAA-680 / CLIP 11262 |
| 分子量 | 理論値: 31.864547 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP ...文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP LIGTID(TPO)ERA KLIIENLLIG VVKNRSEVVL IDITGVPVVD TMVAHHIIQA SEAVRLVGCQ AMLVGIRP E IAQ(TPO)IVNLGI ELDQIITTNT MKKGMERALA LTNREIVEKE G UniProtKB: RsbR protein |
-分子 #5: RsbR protein
| 分子 | 名称: RsbR protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア) Listeria innocua serovar 6a (strain ATCC BAA-680 / CLIP 11262) (バクテリア)株: ATCC BAA-680 / CLIP 11262 |
| 分子量 | 理論値: 31.784566 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP ...文字列: MYKDFANFIR TNKKDLLNNW MNEMEKQSDP LINDIAKEPM YEETSIEFVD LIVSNITENG SKFNEKLDDF AEKVVHLGWP IHFVTTGLR VFGLLVYTAM RDEDLFLKRE EKPEDDAYYR FETWLSSMYN KVVTAYADTW EKTVSIQKSA LQELSAPLLP I FEKISVMP LIGTID(TPO)ERA KLIIENLLIG VVKNRSEVVL IDITGVPVVD TMVAHHIIQA SEAVRLVGCQ AMLVGIRP E IAQTIVNLGI ELDQIITTNT MKKGMERALA LTNREIVEKE G UniProtKB: RsbR protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.5 構成要素:
詳細: pH 8.5 | |||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 気圧: 0.029 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: D2 (2回x2回 2面回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.86 Å / 解像度の算出法: FSC 0.143 CUT-OFF ソフトウェア: (名称: RELION (ver. 3.0), cryoSPARC (ver. V2)) 使用した粒子像数: 32031 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-7b0u: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)





















