+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11514 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Molecular pore spanning the double membranes of murine coronaviral (MHV-A59) replication organelles. | |||||||||
 マップデータ マップデータ | In situ structure of the molecular pore that spans the double membranes of coronavirus (MHV-A59) induced replication organelles. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Murine hepatitis virus strain A59 (ウイルス) Murine hepatitis virus strain A59 (ウイルス) | |||||||||
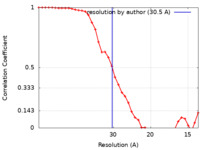
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 30.5 Å | |||||||||
 データ登録者 データ登録者 | Barcena M / Wolff G | |||||||||
| 資金援助 |  オランダ, 1件 オランダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: A molecular pore spans the double membrane of the coronavirus replication organelle. 著者: Georg Wolff / Ronald W A L Limpens / Jessika C Zevenhoven-Dobbe / Ulrike Laugks / Shawn Zheng / Anja W M de Jong / Roman I Koning / David A Agard / Kay Grünewald / Abraham J Koster / Eric J ...著者: Georg Wolff / Ronald W A L Limpens / Jessika C Zevenhoven-Dobbe / Ulrike Laugks / Shawn Zheng / Anja W M de Jong / Roman I Koning / David A Agard / Kay Grünewald / Abraham J Koster / Eric J Snijder / Montserrat Bárcena /    要旨: Coronavirus genome replication is associated with virus-induced cytosolic double-membrane vesicles, which may provide a tailored microenvironment for viral RNA synthesis in the infected cell. ...Coronavirus genome replication is associated with virus-induced cytosolic double-membrane vesicles, which may provide a tailored microenvironment for viral RNA synthesis in the infected cell. However, it is unclear how newly synthesized genomes and messenger RNAs can travel from these sealed replication compartments to the cytosol to ensure their translation and the assembly of progeny virions. In this study, we used cellular cryo-electron microscopy to visualize a molecular pore complex that spans both membranes of the double-membrane vesicle and would allow export of RNA to the cytosol. A hexameric assembly of a large viral transmembrane protein was found to form the core of the crown-shaped complex. This coronavirus-specific structure likely plays a key role in coronavirus replication and thus constitutes a potential drug target. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11514.map.gz emd_11514.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11514-v30.xml emd-11514-v30.xml emd-11514.xml emd-11514.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11514_fsc.xml emd_11514_fsc.xml | 4.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11514.png emd_11514.png | 60.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11514 http://ftp.pdbj.org/pub/emdb/structures/EMD-11514 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11514 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11514 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11514_validation.pdf.gz emd_11514_validation.pdf.gz | 252 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11514_full_validation.pdf.gz emd_11514_full_validation.pdf.gz | 251.1 KB | 表示 | |
| XML形式データ |  emd_11514_validation.xml.gz emd_11514_validation.xml.gz | 7.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11514 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11514 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11514 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11514 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11514.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11514.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | In situ structure of the molecular pore that spans the double membranes of coronavirus (MHV-A59) induced replication organelles. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.02 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Molecular pore containing hexameric nsp3 in the double membranes ...
| 全体 | 名称: Molecular pore containing hexameric nsp3 in the double membranes of the replication organelles induced by murine coronavirus. |
|---|---|
| 要素 |
|
-超分子 #1: Molecular pore containing hexameric nsp3 in the double membranes ...
| 超分子 | 名称: Molecular pore containing hexameric nsp3 in the double membranes of the replication organelles induced by murine coronavirus. タイプ: complex / ID: 1 / 親要素: 0 詳細: Upon infection with murine coronavirus (MHV-A59) the formation of ER derived double-membrane vesicles is induced in the host cell. The molecular pore spans the two membranes of these viral replication organelles. |
|---|---|
| 由来(天然) | 生物種:  Murine hepatitis virus strain A59 (ウイルス) / Organelle: virus-induced double-membrane vesicles / 細胞中の位置: perinuclear area Murine hepatitis virus strain A59 (ウイルス) / Organelle: virus-induced double-membrane vesicles / 細胞中の位置: perinuclear area |
| 組換発現 | 生物種:  Murine hepatitis virus strain A59 (ウイルス) Murine hepatitis virus strain A59 (ウイルス) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 310 K / 装置: LEICA EM GP / 詳細: Blotting time 15s. |
| 詳細 | electron cryo-tomography of FIB-milled lamellae |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 2.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)