+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1132 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A partial atomic structure for the flagellar hook of Salmonella typhimurium. | |||||||||
 マップデータ マップデータ | This is a map of the Salmonell Typhimurium Flagellar hook | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bacterial-type flagellum hook / bacterial-type flagellum basal body / bacterial-type flagellum-dependent swarming motility / bacterial-type flagellum / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 9.0 Å | |||||||||
 データ登録者 データ登録者 | Shaikh TR / Thomas DR / Chen JZ / Samatey FA / Matsunami H / Imada K / Namba K / DeRosier DJ | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2005 ジャーナル: Proc Natl Acad Sci U S A / 年: 2005タイトル: A partial atomic structure for the flagellar hook of Salmonella typhimurium. 著者: Tanvir R Shaikh / Dennis R Thomas / James Z Chen / Fadel A Samatey / Hideyuki Matsunami / Katsumi Imada / Keiichi Namba / David J Derosier /  要旨: The axial proteins of the bacterial flagellum function as a drive shaft, universal joint, and propeller driven by the flagellar rotary motor; they also form the putative protein export channel. The N- ...The axial proteins of the bacterial flagellum function as a drive shaft, universal joint, and propeller driven by the flagellar rotary motor; they also form the putative protein export channel. The N- and C-terminal sequences of the eight axial proteins were predicted to form interlocking alpha-domains generating an axial tube. We report on an approximately 1-nm resolution map of the hook from Salmonella typhimurium, which reveals such a tube made from interdigitated, 1-nm rod-like densities similar to those seen in maps of the filament. Atomic models for the two outer domains of the hook subunit were docked into the corresponding outermost features of the map. The N and C termini of the hook subunit fragment are positioned next to each other and face toward the axis of the hook. The placement of these termini would permit the residues missing in the fragment to form the rod-like features that form the core domain of the hook. We also fit the hook atomic model to an approximately 2-nm resolution map of the hook from Caulobacter crescentus. The hook protein sequence from C. crescentus is largely homologous to that of S. typhimurium except for a large insertion (20 kDa). According to difference maps and our fitting, this insertion is found on the outer surface of the hook, consistent with our modeling of the hook. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1132.map.gz emd_1132.map.gz | 3.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1132-v30.xml emd-1132-v30.xml emd-1132.xml emd-1132.xml | 10.8 KB 10.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1132.gif 1132.gif | 17.6 KB | ||
| Filedesc layerLines |  emd_1132_ll.cif.gz emd_1132_ll.cif.gz | 23.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1132 http://ftp.pdbj.org/pub/emdb/structures/EMD-1132 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1132 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1132 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1132_validation.pdf.gz emd_1132_validation.pdf.gz | 231.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1132_full_validation.pdf.gz emd_1132_full_validation.pdf.gz | 230.2 KB | 表示 | |
| XML形式データ |  emd_1132_validation.xml.gz emd_1132_validation.xml.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1132 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1132 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1132 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1132 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1132.map.gz / 形式: CCP4 / 大きさ: 4.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1132.map.gz / 形式: CCP4 / 大きさ: 4.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a map of the Salmonell Typhimurium Flagellar hook | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.18 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
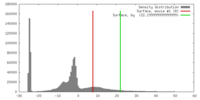
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Salmonella typhimurium Flagellar hook
| 全体 | 名称: Salmonella typhimurium Flagellar hook |
|---|---|
| 要素 |
|
-超分子 #1000: Salmonella typhimurium Flagellar hook
| 超分子 | 名称: Salmonella typhimurium Flagellar hook / タイプ: sample / ID: 1000 詳細: sample is derived from a polyhook mutant in the hook length controling gene fliK 集合状態: Segment of helix containing 75 hooksubunits / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 3.1 MDa / 手法: amino acid sequence |
-分子 #1: flagellar hook protein
| 分子 | 名称: flagellar hook protein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: FlgE / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌)株: SJW880 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 10 mM tris, 5 mM EDTA, 0.1% triton X-100 |
|---|---|
| グリッド | 詳細: 300 mesh copper grids with holey carbon films |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: gravity plunger. vitrification done in cold room at 4 deg C. Polyhooks are straight at 4 deg. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 温度 | 平均: 100 K |
| アライメント法 | Legacy - 非点収差: corrected at 300,000 |
| 撮影 | デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 21 µm / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 66000 |
| 試料ステージ | 試料ホルダー: cryo / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | Hook is naturally helical when assembled |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: BRANDEIS HELICAL PACKAGE 詳細: average includes 262 datasets and included 46 layerlines out of 84 collected. |
| CTF補正 | 詳細: corrected averaged layerlines |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: RSRef |
| 詳細 | PDBEntryID_givenInChain. Protocol: semi-rigid body. Two domains are kept as independent rigid bodies, connected by flexible peptide. Refinement is via simulated annealing molecular dynamics, including the adjacent subunits in the helix. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: correl. coeff. |
| 得られたモデル |  PDB-2bgy:  PDB-2bgz: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)