+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11078 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of the matrix protein M1 from influenza A/Hong Kong/1/1968 VLPs (HA,NA,M1,M2) | ||||||||||||
 マップデータ マップデータ | Structure of the influenza A matrix protein M1 from influenza A/Hong Kong/1/1968 VLPs (HA,NA,M1,M2) | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Viral matrix protein / Membrane binding / pH Sensor / Polymer / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virion assembly / viral budding from plasma membrane / structural constituent of virion / host cell nucleus / virion membrane / RNA binding / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /  Influenza A virus (A/Puerto Rico/8-9NMC3/1934(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Puerto Rico/8-9NMC3/1934(H1N1)) (A型インフルエンザウイルス) | ||||||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 8.0 Å | ||||||||||||
 データ登録者 データ登録者 | Peukes J / Xiong X | ||||||||||||
| 資金援助 | European Union,  英国, 英国,  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: The native structure of the assembled matrix protein 1 of influenza A virus. 著者: Julia Peukes / Xiaoli Xiong / Simon Erlendsson / Kun Qu / William Wan / Leslie J Calder / Oliver Schraidt / Susann Kummer / Stefan M V Freund / Hans-Georg Kräusslich / John A G Briggs /      要旨: Influenza A virus causes millions of severe cases of disease during annual epidemics. The most abundant protein in influenza virions is matrix protein 1 (M1), which mediates virus assembly by ...Influenza A virus causes millions of severe cases of disease during annual epidemics. The most abundant protein in influenza virions is matrix protein 1 (M1), which mediates virus assembly by forming an endoskeleton beneath the virus membrane. The structure of full-length M1, and how it oligomerizes to mediate the assembly of virions, is unknown. Here we determine the complete structure of assembled M1 within intact virus particles, as well as the structure of M1 oligomers reconstituted in vitro. We find that the C-terminal domain of M1 is disordered in solution but can fold and bind in trans to the N-terminal domain of another M1 monomer, thus polymerizing M1 into linear strands that coat the interior surface of the membrane of the assembling virion. In the M1 polymer, five histidine residues-contributed by three different monomers of M1-form a cluster that can serve as the pH-sensitive disassembly switch after entry into a target cell. These structures therefore reveal mechanisms of influenza virus assembly and disassembly. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11078.map.gz emd_11078.map.gz | 6.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11078-v30.xml emd-11078-v30.xml emd-11078.xml emd-11078.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11078_fsc.xml emd_11078_fsc.xml | 4.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11078.png emd_11078.png | 137.9 KB | ||
| マスクデータ |  emd_11078_msk_1.map emd_11078_msk_1.map | 6.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11078.cif.gz emd-11078.cif.gz | 6.8 KB | ||
| その他 |  emd_11078_half_map_1.map.gz emd_11078_half_map_1.map.gz emd_11078_half_map_2.map.gz emd_11078_half_map_2.map.gz | 6.1 MB 6.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11078 http://ftp.pdbj.org/pub/emdb/structures/EMD-11078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11078 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11078_validation.pdf.gz emd_11078_validation.pdf.gz | 581.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11078_full_validation.pdf.gz emd_11078_full_validation.pdf.gz | 580.6 KB | 表示 | |
| XML形式データ |  emd_11078_validation.xml.gz emd_11078_validation.xml.gz | 10 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11078 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11078 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11078.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11078.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the influenza A matrix protein M1 from influenza A/Hong Kong/1/1968 VLPs (HA,NA,M1,M2) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.78 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11078_msk_1.map emd_11078_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
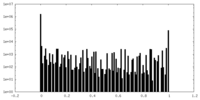
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11078_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
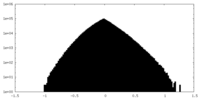
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11078_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
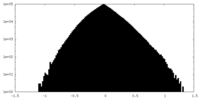
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Influenza A virus
| 全体 | 名称:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Influenza A virus
| 超分子 | 名称: Influenza A virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: virus like particles were formed by expressing plasmids coding for a subset (HA,NA,M1,M2) of the viral proteins NCBI-ID: 11320 / 生物種: Influenza A virus / Sci species strain: A/Hong Kong/1/1968 (H3N2) / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: Matrix protein 1
| 超分子 | 名称: Matrix protein 1 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 詳細: The M1 map was generated by subtomogram averaging of the M1 protein density in tomograms of influenza A (HK68) virus like particles (VLPs) that were generated by expression of the viral protein HA,NA,M1,M2 |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス)株: A/Hong Kong/1/1968 (H3N2) |
-分子 #1: Matrix protein 1
| 分子 | 名称: Matrix protein 1 / タイプ: protein_or_peptide / ID: 1 詳細: The PDB model was generated by rigid body fitting of the M1 NTD crystal structure (PBD: 1ea3, from PR8 influenza virus) into the EM density map obtained for HK68 VLPs. コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Puerto Rico/8-9NMC3/1934(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Puerto Rico/8-9NMC3/1934(H1N1)) (A型インフルエンザウイルス)株: A/Puerto Rico/8-9NMC3/1934(H1N1) |
| 分子量 | 理論値: 27.928301 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSLLTEVETY VLSIIPSGPL KAEIAQRLED VFAGKNTDLE VLMEWLKTRP ILSPLTKGIL GFVFTLTVPS ERGLQRRRFV QNALNGNGD PNNMDKAVKL YRKLKREITF HGAKEISLSY SAGALASCMG LIYNRMGAVT TEVAFGLVCA TCEQIADSQH R SHRQMVTT ...文字列: MSLLTEVETY VLSIIPSGPL KAEIAQRLED VFAGKNTDLE VLMEWLKTRP ILSPLTKGIL GFVFTLTVPS ERGLQRRRFV QNALNGNGD PNNMDKAVKL YRKLKREITF HGAKEISLSY SAGALASCMG LIYNRMGAVT TEVAFGLVCA TCEQIADSQH R SHRQMVTT TNPLIRHENR MVLASTTAKA MEQMAGSSEQ AAEAMEVASQ ARQMVQAMRT IGTHPSSSAG LKNDLLENLQ AY QKRMGVQ MQRFK UniProtKB: Matrix protein 1 |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 258 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 2.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Multiple copies of the M1 NTD crystal structure (PDB:1ea3) were fitted as rigid bodies into the EM map to understand the relative arrangement of M1 monomers in the context of the matrix layer inside the virus |
| 精密化 | プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-6z5j: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)