+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10998 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mitochondrial ATP synthase monomer from Rattus norvegicus domestica | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
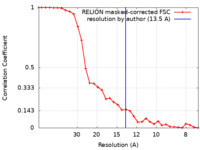
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 13.5 Å | |||||||||
 データ登録者 データ登録者 | Chesnokov YM / Kamyshinsky RA | |||||||||
 引用 引用 |  ジャーナル: Nanotechnol Russia / 年: 2020 ジャーナル: Nanotechnol Russia / 年: 2020タイトル: Determining the Structure and Location of the ATP Synthase in the Membranes of Rat's Heart Mitochondria Using Cryoelectron Tomography 著者: Nesterov SV / Chesnokov YM / Kamyshinsky RA / Yaguzhinsky LS / Vasilov RG | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10998.map.gz emd_10998.map.gz | 682.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10998-v30.xml emd-10998-v30.xml emd-10998.xml emd-10998.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10998_fsc.xml emd_10998_fsc.xml | 3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10998.png emd_10998.png | 34.9 KB | ||
| マスクデータ |  emd_10998_msk_1.map emd_10998_msk_1.map | 2 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10998_half_map_1.map.gz emd_10998_half_map_1.map.gz emd_10998_half_map_2.map.gz emd_10998_half_map_2.map.gz | 1.4 MB 1.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10998 http://ftp.pdbj.org/pub/emdb/structures/EMD-10998 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10998 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10998 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10998_validation.pdf.gz emd_10998_validation.pdf.gz | 314.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10998_full_validation.pdf.gz emd_10998_full_validation.pdf.gz | 314.1 KB | 表示 | |
| XML形式データ |  emd_10998_validation.xml.gz emd_10998_validation.xml.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10998 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10998 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10998 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10998 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  10997 |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10998.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10998.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
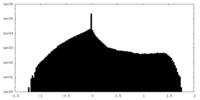
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10998_msk_1.map emd_10998_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
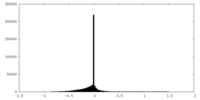
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10998_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
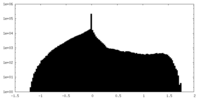
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10998_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
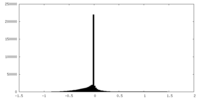
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mitochondrial ATP synthase dimer from Rattus norvegicus
| 全体 | 名称: Mitochondrial ATP synthase dimer from Rattus norvegicus |
|---|---|
| 要素 |
|
-超分子 #1: Mitochondrial ATP synthase dimer from Rattus norvegicus
| 超分子 | 名称: Mitochondrial ATP synthase dimer from Rattus norvegicus タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 600 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 2.5 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: Cs image corrector (CEOS, Germany) |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 平均露光時間: 1.5 sec. / 平均電子線量: 1.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 37837 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 6.0 µm / 倍率(公称値): 18000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)