+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10765 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron tomogram and segmentation of the cortical ER in yeast used for testing membrane curvature estimation algorithms. | ||||||||||||
 マップデータ マップデータ | Raw tomogram. | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | ||||||||||||
 データ登録者 データ登録者 | Collado JF / Salfer M | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: PLoS Comput Biol / 年: 2020 ジャーナル: PLoS Comput Biol / 年: 2020タイトル: Reliable estimation of membrane curvature for cryo-electron tomography. 著者: Maria Salfer / Javier F Collado / Wolfgang Baumeister / Rubén Fernández-Busnadiego / Antonio Martínez-Sánchez /  要旨: Curvature is a fundamental morphological descriptor of cellular membranes. Cryo-electron tomography (cryo-ET) is particularly well-suited to visualize and analyze membrane morphology in a close-to- ...Curvature is a fundamental morphological descriptor of cellular membranes. Cryo-electron tomography (cryo-ET) is particularly well-suited to visualize and analyze membrane morphology in a close-to-native state and molecular resolution. However, current curvature estimation methods cannot be applied directly to membrane segmentations in cryo-ET, as these methods cannot cope with some of the artifacts introduced during image acquisition and membrane segmentation, such as quantization noise and open borders. Here, we developed and implemented a Python package for membrane curvature estimation from tomogram segmentations, which we named PyCurv. From a membrane segmentation, a signed surface (triangle mesh) is first extracted. The triangle mesh is then represented by a graph, which facilitates finding neighboring triangles and the calculation of geodesic distances necessary for local curvature estimation. PyCurv estimates curvature based on tensor voting. Beside curvatures, this algorithm also provides robust estimations of surface normals and principal directions. We tested PyCurv and three well-established methods on benchmark surfaces and biological data. This revealed the superior performance of PyCurv not only for cryo-ET, but also for data generated by other techniques such as light microscopy and magnetic resonance imaging. Altogether, PyCurv is a versatile open-source software to reliably estimate curvature of membranes and other surfaces in a wide variety of applications. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10765.map.gz emd_10765.map.gz | 91.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10765-v30.xml emd-10765-v30.xml emd-10765.xml emd-10765.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10765.png emd_10765.png | 239.7 KB | ||
| マスクデータ |  emd_10765_msk_1.map emd_10765_msk_1.map | 12.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10765_additional.map.gz emd_10765_additional.map.gz emd_10765_additional_1.map.gz emd_10765_additional_1.map.gz | 113.1 MB 113.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10765 http://ftp.pdbj.org/pub/emdb/structures/EMD-10765 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10765 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10765 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10765_validation.pdf.gz emd_10765_validation.pdf.gz | 168.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10765_full_validation.pdf.gz emd_10765_full_validation.pdf.gz | 167.5 KB | 表示 | |
| XML形式データ |  emd_10765_validation.xml.gz emd_10765_validation.xml.gz | 4.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10765 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10765 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10765 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10765 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10765.map.gz / 形式: CCP4 / 大きさ: 202 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_10765.map.gz / 形式: CCP4 / 大きさ: 202 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw tomogram. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 13.68 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10765_msk_1.map emd_10765_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
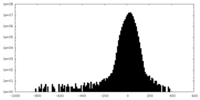
| 密度ヒストグラム |
-追加マップ: Filtered tomogram using a deconvolution filter (https://github.com/dtegunov/tom deconv) executed...
| ファイル | emd_10765_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Filtered tomogram using a deconvolution filter (https://github.com/dtegunov/tom_deconv) executed in MATLAB (Mathworks) using the functionalities of the TOM toolbox (Nickel et al. 2005). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
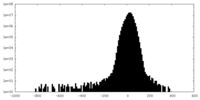
| 密度ヒストグラム |
-追加マップ: Filtered tomogram using a deconvolution filter (https://github.com/dtegunov/tom deconv) executed...
| ファイル | emd_10765_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Filtered tomogram using a deconvolution filter (https://github.com/dtegunov/tom_deconv) executed in MATLAB (Mathworks) using the functionalities of the TOM toolbox (Nickel et al. 2005). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Saccharomyces cerevisiae strain ANDY129 delta Scs2 delta Scs22 de...
| 全体 | 名称: Saccharomyces cerevisiae strain ANDY129 delta Scs2 delta Scs22 delta Ist2 |
|---|---|
| 要素 |
|
-超分子 #1: Saccharomyces cerevisiae strain ANDY129 delta Scs2 delta Scs22 de...
| 超分子 | 名称: Saccharomyces cerevisiae strain ANDY129 delta Scs2 delta Scs22 delta Ist2 タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 切片作成 | 集束イオンビーム - 装置: OTHER / 集束イオンビーム - イオン: OTHER / 集束イオンビーム - 電圧: 30 kV / 集束イオンビーム - 電流: 0.03 nA / 集束イオンビーム - 時間: 4000 sec. / 集束イオンビーム - 温度: 93 K / 集束イオンビーム - Initial thickness: 1000 nm / 集束イオンビーム - 最終 厚さ: 200 nm 集束イオンビーム - 詳細: The value given for _emd_sectioning_focused_ion_beam.instrument is Quanta 3D cryo-FIB / SEM. This is not in a list of allowed values {'DB235', 'OTHER'} so OTHER is ...集束イオンビーム - 詳細: The value given for _emd_sectioning_focused_ion_beam.instrument is Quanta 3D cryo-FIB / SEM. This is not in a list of allowed values {'DB235', 'OTHER'} so OTHER is written into the XML file. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 90.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 1.6 sec. / 平均電子線量: 1.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.1 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION / ソフトウェア - 名称:  IMOD / 使用した粒子像数: 52 IMOD / 使用した粒子像数: 52 |
|---|
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X