+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1075 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Molecular architecture of the prolate head of bacteriophage T4. | |||||||||
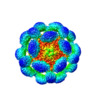
 マップデータ マップデータ | cryoEM reconstruction of the bacteriophage T4 head | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 22.5 Å | |||||||||
 データ登録者 データ登録者 | Fokine A / Chipman PR / Leiman PG / Mesyanzhinov VV / Rao VB / Rossmann MG | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2004 ジャーナル: Proc Natl Acad Sci U S A / 年: 2004タイトル: Molecular architecture of the prolate head of bacteriophage T4. 著者: Andrei Fokine / Paul R Chipman / Petr G Leiman / Vadim V Mesyanzhinov / Venigalla B Rao / Michael G Rossmann /  要旨: The head of bacteriophage T4 is a prolate icosahedron with one unique portal vertex to which the phage tail is attached. The three-dimensional structure of mature bacteriophage T4 head has been ...The head of bacteriophage T4 is a prolate icosahedron with one unique portal vertex to which the phage tail is attached. The three-dimensional structure of mature bacteriophage T4 head has been determined to 22-A resolution by using cryo-electron microscopy. The T4 capsid has a hexagonal surface lattice characterized by the triangulation numbers T(end) = 13 laevo for the icosahedral caps and T(mid) = 20 for the midsection. Hexamers of the major capsid protein gene product (gp)23* and pentamers of the vertex protein gp24*, as well as the outer surface proteins highly antigenic outer capsid protein (hoc) and small outer capsid protein (soc), are clearly evident in the reconstruction. The size and shape of the gp23* hexamers are similar to the major capsid protein organization of bacteriophage HK97. The binding sites and shape of the hoc and soc proteins have been established by analysis of the soc(-) and hoc(-)soc(-) T4 structures. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1075.map.gz emd_1075.map.gz | 22.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1075-v30.xml emd-1075-v30.xml emd-1075.xml emd-1075.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1075.gif 1075.gif 400_1075.gif 400_1075.gif 400_10750.gif 400_10750.gif 400_10751.gif 400_10751.gif 400_10752.gif 400_10752.gif 400_10753.gif 400_10753.gif 400_10754.gif 400_10754.gif 400_10755.gif 400_10755.gif 400_10756.gif 400_10756.gif 400_10757.gif 400_10757.gif 400_10758.gif 400_10758.gif 400_10759.gif 400_10759.gif 80_1075.gif 80_1075.gif 80_10750.gif 80_10750.gif 80_10751.gif 80_10751.gif 80_10752.gif 80_10752.gif 80_10753.gif 80_10753.gif 80_10754.gif 80_10754.gif 80_10755.gif 80_10755.gif 80_10756.gif 80_10756.gif 80_10757.gif 80_10757.gif 80_10758.gif 80_10758.gif 80_10759.gif 80_10759.gif emd_1075.tif emd_1075.tif | 74.2 KB 61.4 KB 62.7 KB 55.9 KB 37.8 KB 29.4 KB 90.3 KB 106.2 KB 72.4 KB 51.9 KB 104.2 KB 76.2 KB 2.7 KB 4.3 KB 3.7 KB 2.9 KB 2.8 KB 5.4 KB 5.9 KB 4.5 KB 3.5 KB 5.9 KB 4.8 KB 737.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1075 http://ftp.pdbj.org/pub/emdb/structures/EMD-1075 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1075 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1075 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1075_validation.pdf.gz emd_1075_validation.pdf.gz | 239.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1075_full_validation.pdf.gz emd_1075_full_validation.pdf.gz | 238.5 KB | 表示 | |
| XML形式データ |  emd_1075_validation.xml.gz emd_1075_validation.xml.gz | 4.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1075 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1075 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1075 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1075 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1075.map.gz / 形式: CCP4 / 大きさ: 27.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1075.map.gz / 形式: CCP4 / 大きさ: 27.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM reconstruction of the bacteriophage T4 head | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.96 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : bacteriophage T4 prolate head
| 全体 | 名称: bacteriophage T4 prolate head |
|---|---|
| 要素 |
|
-超分子 #1000: bacteriophage T4 prolate head
| 超分子 | 名称: bacteriophage T4 prolate head / タイプ: sample / ID: 1000 詳細: Sample was the native bacteriophage T4. Only the head part of the phage was reconstructed. Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 194 MDa / 手法: STEM, 194 MDa is the mass of the DNA filled head. |
-超分子 #1: Enterobacteria phage T4
| 超分子 | 名称: Enterobacteria phage T4 / タイプ: virus / ID: 1 / Name.synonym: phage T4 / NCBI-ID: 10665 / 生物種: Enterobacteria phage T4 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: phage T4 |
|---|---|
| 宿主 | 生物種: Ecoli / 別称: BACTERIA(EUBACTERIA) |
| 分子量 | 理論値: 194 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: capsid width is 860 A, length is 1195 A |
| ウイルス殻 | Shell ID: 2 / 名称: Tmid=20; Tend=13 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.2 詳細: Distilled water. The concentration of the virus particles was 10E11 particles pre ml. |
|---|---|
| グリッド | 詳細: quantifoil copper grid 2um holes |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 90 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: in-house, gravity driven plunger 手法: 3.5ul of sample hand blotted approx. 1sec |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM300FEG/T |
|---|---|
| 温度 | 平均: 97 K |
| アライメント法 | Legacy - 非点収差: live fft at 190,000x |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 55 / 平均電子線量: 29 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 47000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.1 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C5 (5回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 22.5 Å / 解像度の算出法: FSC 0.33 CUT-OFF / ソフトウェア - 名称: SPIDER 詳細: The reconstruction was performed by imposing 5-fold symmetry averaging. The 5-fold axis is along Z. 使用した粒子像数: 5140 |
| 最終 角度割当 | 詳細: The reconstruction was made in SPIDER using the projection matching protocol. The reference projections were calculated in the interval theta: 0-90 degrees, phi: 0-72 degrees. |
-原子モデル構築 1
| 詳細 | Nothing to fit |
|---|
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















