+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1072 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structures of translating ribosomes by Cryo-EM. | |||||||||
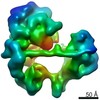
 マップデータ マップデータ | 3D reconstruction of E. coli ribosome stalled in translation of Green Fluorescent Protein | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Gilbert RJC / Fucini P / Connell S / Fuller SD / Nierhaus KH / Robinson CV / Dobson CM / Stuart DI | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2004 ジャーナル: Mol Cell / 年: 2004タイトル: Three-dimensional structures of translating ribosomes by Cryo-EM. 著者: Robert J C Gilbert / Paola Fucini / Sean Connell / Stephen D Fuller / Knud H Nierhaus / Carol V Robinson / Christopher M Dobson / David I Stuart /  要旨: Cryo-electron microscopy and image reconstruction techniques have been used to obtain three-dimensional maps for E. coli ribosomes stalled following translation of three representative proteins. ...Cryo-electron microscopy and image reconstruction techniques have been used to obtain three-dimensional maps for E. coli ribosomes stalled following translation of three representative proteins. Comparisons of these electron density maps, at resolutions of between 13 and 16 A, with that of a nontranslating ribosome pinpoint specific structural differences in stalled ribosomes and identify additional material, including tRNAs and mRNA. In addition, the tunnel through the large subunit, the anticipated exit route of newly synthesized proteins, is partially occluded in all the stalled ribosome structures. This observation suggests that significant segments of the nascent polypeptide chains examined here could be located within an expanded tunnel, perhaps in a rudimentary globular conformation. Such behavior could be an important aspect of the folding of at least some proteins in the cellular environment. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1072.map.gz emd_1072.map.gz | 7.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1072-v30.xml emd-1072-v30.xml emd-1072.xml emd-1072.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1072.gif 1072.gif | 61.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1072 http://ftp.pdbj.org/pub/emdb/structures/EMD-1072 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1072 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1072 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1072_validation.pdf.gz emd_1072_validation.pdf.gz | 236.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1072_full_validation.pdf.gz emd_1072_full_validation.pdf.gz | 235.5 KB | 表示 | |
| XML形式データ |  emd_1072_validation.xml.gz emd_1072_validation.xml.gz | 5.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1072 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1072 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1072 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1072 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1072.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1072.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of E. coli ribosome stalled in translation of Green Fluorescent Protein | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.33 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E. coli ribosome translating GFP
| 全体 | 名称: E. coli ribosome translating GFP |
|---|---|
| 要素 |
|
-超分子 #1000: E. coli ribosome translating GFP
| 超分子 | 名称: E. coli ribosome translating GFP / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse. 集合状態: 30S subunit and 50S subunit, 2 tRNA molecules and 1 nascent protein Number unique components: 5 |
|---|
-超分子 #1: E. coli 30S subunit
| 超分子 | 名称: E. coli 30S subunit / タイプ: complex / ID: 1 / Name.synonym: 30S / 組換発現: No / Ribosome-details: ribosome-prokaryote: SSU 30S |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: E. coli 50S subunit
| 超分子 | 名称: E. coli 50S subunit / タイプ: complex / ID: 2 / Name.synonym: 50S / 組換発現: No / Ribosome-details: ribosome-prokaryote: LSU 50S |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: P site tRNA
| 分子 | 名称: P site tRNA / タイプ: rna / ID: 1 / 詳細: partial occupancy / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #2: E site tRNA
| 分子 | 名称: E site tRNA / タイプ: rna / ID: 2 / 詳細: partial occupancy lower than P site / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #3: Green fluorescent protein
| 分子 | 名称: Green fluorescent protein / タイプ: protein_or_peptide / ID: 3 / Name.synonym: GFP / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種: In vitro transcription.translation system / 組換プラスミド: pIVEX2.3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.0 mg/mL |
|---|---|
| 緩衝液 | 詳細: 20 mM HEPES, 150 mM ammonium acetate, 6 mM magnesium acetate, 2 mM spermidine, 0.05 mM spermine and 4 mM 2-mercaptoethanol. Concentration of ribosomes expressed to A260 units. |
| グリッド | 詳細: 300 mesh copper grid with holey carbon film |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 100 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: Standard unmodified guillotine plunger |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 温度 | 平均: 100 K |
| 日付 | 2000年5月2日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 8.322 µm / 実像数: 10 / 詳細: Scanner model:UMAX PowerLook 3000 / Od range: 5 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / Cs: 2 mm / 最大 デフォーカス(公称値): 5.96 µm / 最小 デフォーカス(公称値): 1.805 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each negative dataset |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF ソフトウェア - 名称: SPIDER, IMAGIC, GAP, CNS, XPLOR 詳細: Final maps were calculated from 2276 images from a total of 4500 from 10 individual datasets, with scaling in reciprocal space to crystallographic ribosome structures and correction for map ...詳細: Final maps were calculated from 2276 images from a total of 4500 from 10 individual datasets, with scaling in reciprocal space to crystallographic ribosome structures and correction for map anisotropy by B-factor weighting of amplitudes in XPLOR with respect to a similarly-treated control inactive ribosome. Selection of particles for inclusion in the final maps was by correlation coefficient with respect to the alignment model, designed to maximise nascent chain occupancy in the selected images. 使用した粒子像数: 2276 |
| 最終 角度割当 | 詳細: SPIDER euler |
-原子モデル構築 1
| ソフトウェア | 名称: GAP |
|---|---|
| 詳細 | Protocol: Rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: R-factor and correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー














 Y (Sec.)
Y (Sec.) X (Row.)
X (Row.) Z (Col.)
Z (Col.)





















