+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10561 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the RBM3/collar region of the Salmonella flagella MS-ring protein FliF with 35-fold symmetry applied | |||||||||||||||||||||
 マップデータ マップデータ | Post process map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 生物種 |  Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) | |||||||||||||||||||||
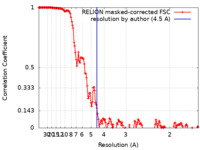
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Johnson S / Fong YH / Deme JC / Furlong EJ / Kuhlen L / Lea SM | |||||||||||||||||||||
| 資金援助 |  英国, 6件 英国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2020 ジャーナル: Nat Microbiol / 年: 2020タイトル: Symmetry mismatch in the MS-ring of the bacterial flagellar rotor explains the structural coordination of secretion and rotation. 著者: Steven Johnson / Yu Hang Fong / Justin C Deme / Emily J Furlong / Lucas Kuhlen / Susan M Lea /  要旨: The bacterial flagellum is a complex self-assembling nanomachine that confers motility to the cell. Despite great variation across species, all flagella are ultimately constructed from a helical ...The bacterial flagellum is a complex self-assembling nanomachine that confers motility to the cell. Despite great variation across species, all flagella are ultimately constructed from a helical propeller that is attached to a motor embedded in the inner membrane. The motor consists of a series of stator units surrounding a central rotor made up of two ring complexes, the MS-ring and the C-ring. Despite many studies, high-resolution structural information is still lacking for the MS-ring of the rotor, and proposed mismatches in stoichiometry between the two rings have long provided a source of confusion for the field. Here, we present structures of the Salmonella MS-ring, revealing a high level of variation in inter- and intrachain symmetry that provides a structural explanation for the ability of the MS-ring to function as a complex and elegant interface between the two main functions of the flagellum-protein secretion and rotation. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10561.map.gz emd_10561.map.gz | 36.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10561-v30.xml emd-10561-v30.xml emd-10561.xml emd-10561.xml | 18.9 KB 18.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10561_fsc.xml emd_10561_fsc.xml | 15.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10561.png emd_10561.png | 46.7 KB | ||
| マスクデータ |  emd_10561_msk_1.map emd_10561_msk_1.map | 307.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10561_additional.map.gz emd_10561_additional.map.gz emd_10561_additional_1.map.gz emd_10561_additional_1.map.gz emd_10561_half_map_1.map.gz emd_10561_half_map_1.map.gz emd_10561_half_map_2.map.gz emd_10561_half_map_2.map.gz | 237.9 MB 237.9 MB 243.5 MB 243.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10561 http://ftp.pdbj.org/pub/emdb/structures/EMD-10561 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10561 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10561 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10561_validation.pdf.gz emd_10561_validation.pdf.gz | 405.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10561_full_validation.pdf.gz emd_10561_full_validation.pdf.gz | 404.7 KB | 表示 | |
| XML形式データ |  emd_10561_validation.xml.gz emd_10561_validation.xml.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10561 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10561 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10561 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10561 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10561.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10561.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post process map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.822 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10561_msk_1.map emd_10561_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Refinement map
| ファイル | emd_10561_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Refinement map
| ファイル | emd_10561_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_10561_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_10561_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homomeric 35mer of Salmonella enterica serovar Typhimurium FliF
| 全体 | 名称: Homomeric 35mer of Salmonella enterica serovar Typhimurium FliF |
|---|---|
| 要素 |
|
-超分子 #1: Homomeric 35mer of Salmonella enterica serovar Typhimurium FliF
| 超分子 | 名称: Homomeric 35mer of Salmonella enterica serovar Typhimurium FliF タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 2.14 MDa |
-分子 #1: Flagellar MS-ring FliF
| 分子 | 名称: Flagellar MS-ring FliF / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSATASTATQ PKPLEWLNRL RANPRIPLIV AGSAAVAIVV AMVLWAKTPD YRTLFSNLSD QDGGAIVAQ LTQMNIPYRF ANGSGAIEVP ADKVHELRLR LAQQGLPKGG AVGFELLDQE K FGISQFSE QVNYQRALEG ELARTIETLG PVKSARVHLA MPKPSLFVRE ...文字列: MSATASTATQ PKPLEWLNRL RANPRIPLIV AGSAAVAIVV AMVLWAKTPD YRTLFSNLSD QDGGAIVAQ LTQMNIPYRF ANGSGAIEVP ADKVHELRLR LAQQGLPKGG AVGFELLDQE K FGISQFSE QVNYQRALEG ELARTIETLG PVKSARVHLA MPKPSLFVRE QKSPSASVTV TL EPGRALD EGQISAVVHL VSSAVAGLPP GNVTLVDQSG HLLTQSNTSG RDLNDAQLKF AND VESRIQ RRIEAILSPI VGNGNVHAQV TAQLDFANKE QTEEHYSPNG DASKATLRSR QLNI SEQVG AGYPGGVPGA LSNQPAPPNE APIATPPTNQ QNAQNTPQTS TSTNSNSAGP RSTQR NETS NYEVDRTIRH TKMNVGDIER LSVAVVVNYK TLADGKPLPL TADQMKQIED LTREAM GFS DKRGDTLNVV NSPFSAVDNT GGELPFWQQQ SFIDQLLAAG RWLLVLVVAW ILWRKAV RP QLTRRVEEAK AAQEQAQVRQ ETEEAVEVRL SKDEQLQQRR ANQRLGAEVM SQRIREMS D NDPRVVALVI RQWMSNDHE |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)