+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10399 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | FtsK motor domain with dsDNA, translocating state | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA translocation / DNA motor / RecA fold / Divisome / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA translocase activity / cellular response to antibiotic / chromosome segregation / cell division / DNA binding / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) / synthetic construct (人工物) / Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) / synthetic construct (人工物) /  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) | |||||||||
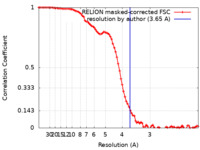
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.65 Å | |||||||||
 データ登録者 データ登録者 | Jean NL / Lowe J | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2020 ジャーナル: Proc Natl Acad Sci U S A / 年: 2020タイトル: FtsK in motion reveals its mechanism for double-stranded DNA translocation. 著者: Nicolas L Jean / Trevor J Rutherford / Jan Löwe /  要旨: FtsK protein contains a fast DNA motor that is involved in bacterial chromosome dimer resolution. During cell division, FtsK translocates double-stranded DNA until both recombination sites are ...FtsK protein contains a fast DNA motor that is involved in bacterial chromosome dimer resolution. During cell division, FtsK translocates double-stranded DNA until both recombination sites are placed at mid cell for subsequent dimer resolution. Here, we solved the 3.6-Å resolution electron cryo-microscopy structure of the motor domain of FtsK while translocating on its DNA substrate. Each subunit of the homo-hexameric ring adopts a unique conformation and one of three nucleotide states. Two DNA-binding loops within four subunits form a pair of spiral staircases within the ring, interacting with the two DNA strands. This suggests that simultaneous conformational changes in all ATPase domains at each catalytic step generate movement through a mechanism related to filament treadmilling. While the ring is only rotating around the DNA slowly, it is instead the conformational states that rotate around the ring as the DNA substrate is pushed through. #1:  ジャーナル: Biorxiv ジャーナル: Biorxivタイトル: FtsK in motion reveals its mechanism for double-stranded DNA translocation 著者: Jean NL / Rutherford TJ / Lowe J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10399.map.gz emd_10399.map.gz | 5.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10399-v30.xml emd-10399-v30.xml emd-10399.xml emd-10399.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10399_fsc.xml emd_10399_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10399.png emd_10399.png | 184.8 KB | ||
| Filedesc metadata |  emd-10399.cif.gz emd-10399.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10399 http://ftp.pdbj.org/pub/emdb/structures/EMD-10399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10399 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10399_validation.pdf.gz emd_10399_validation.pdf.gz | 428.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10399_full_validation.pdf.gz emd_10399_full_validation.pdf.gz | 427.8 KB | 表示 | |
| XML形式データ |  emd_10399_validation.xml.gz emd_10399_validation.xml.gz | 10.5 KB | 表示 | |
| CIF形式データ |  emd_10399_validation.cif.gz emd_10399_validation.cif.gz | 13.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10399 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10399 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10399 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10399 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10399.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10399.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.048 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : FtsK motor domain with dsDNA, translocating state
| 全体 | 名称: FtsK motor domain with dsDNA, translocating state |
|---|---|
| 要素 |
|
-超分子 #1: FtsK motor domain with dsDNA, translocating state
| 超分子 | 名称: FtsK motor domain with dsDNA, translocating state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|
-超分子 #2: FtsK motor domain
| 超分子 | 名称: FtsK motor domain / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: DNA translocase FtsK
| 分子 | 名称: DNA translocase FtsK / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 54.001441 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVPDRREQSK AKERLLEREE ALAKHMSERE KRPPPKIDPP PSPKAPEPSK RVLKEKQAPL FVDTAVEGTL PPLSLLDPAE VKQKSYSPE SLEAMSRLLE IKLKEFGVEV SVDSVHPGPV ITRFEIQPAA GVKVSRISNL AKDLARSLAV ISVRVVEVIP G KTTVGIEI ...文字列: MVPDRREQSK AKERLLEREE ALAKHMSERE KRPPPKIDPP PSPKAPEPSK RVLKEKQAPL FVDTAVEGTL PPLSLLDPAE VKQKSYSPE SLEAMSRLLE IKLKEFGVEV SVDSVHPGPV ITRFEIQPAA GVKVSRISNL AKDLARSLAV ISVRVVEVIP G KTTVGIEI PNEDRQMVRF SEVLSSPEYD EHKSTVPLAL GHDIGGRPII TDLAKMPHLL VAGTTGSGKS VGVNAMLLSI LF KSTPSEA RLIMIDPKML ELSIYEGIPH LLCPVVTDMK EAANALRWSV AEMERRYRLM AAMGVRNLAG FNRKVKDAEE AGT PLTDPL FRRESPDDEP PQLSTLPTIV VVVDEFADMM MIVGKKVEEL IARIAQKARA AGIHLILATQ RPSVDVITGL IKAN IPTRI AFQVSSKIDS RTILDQGGAE QLLGHGDMLY LPPGTGLPIR VHGAFVSDDE VHRVVEAWKL RGAPDYIEDI LAGVD EGGK LHHHHHH UniProtKB: DNA translocase FtsK |
-分子 #2: dsDNA substrate
| 分子 | 名称: dsDNA substrate / タイプ: dna / ID: 2 詳細: Sequence not determined due to sequence heterogeneity of the protein-bound DNA コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 6.129039 KDa |
| 配列 | 文字列: (DA)(DT)(DA)(DT)(DA)(DT)(DA)(DT)(DA)(DT) (DA)(DT)(DA)(DT)(DA)(DT)(DA)(DT)(DA)(DT) |
-分子 #3: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 3 / コピー数: 3 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 3 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | ||||||||
| 詳細 | 0.7 m/mL FtsK 1.5 uM 45 bp DNA |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 3300 / 平均電子線量: 42.95 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)