+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0826 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Cx31.3/GJC3 connexin hemichannel in the presence of calcium | |||||||||
 マップデータ マップデータ | postprocess_masked map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Gap junction / Hemichannel / Hexamer / ATP release / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報connexin complex / gap junction channel activity / myelination / sensory perception of sound / cell-cell signaling / myelin sheath / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
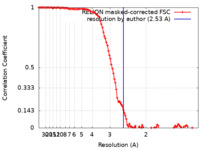
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.53 Å | |||||||||
 データ登録者 データ登録者 | Lee HJ / Jeong H | |||||||||
| 資金援助 |  韓国, 1件 韓国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2020 ジャーナル: Sci Adv / 年: 2020タイトル: Cryo-EM structure of human Cx31.3/GJC3 connexin hemichannel. 著者: Hyuk-Joon Lee / Hyeongseop Jeong / Jaekyung Hyun / Bumhan Ryu / Kunwoong Park / Hyun-Ho Lim / Jejoong Yoo / Jae-Sung Woo /   要旨: Connexin family proteins assemble into hexameric channels called hemichannels/connexons, which function as transmembrane channels or dock together to form gap junction intercellular channels (GJIChs). ...Connexin family proteins assemble into hexameric channels called hemichannels/connexons, which function as transmembrane channels or dock together to form gap junction intercellular channels (GJIChs). We determined the cryo-electron microscopy structures of human connexin 31.3 (Cx31.3)/GJC3 hemichannels in the presence and absence of calcium ions and with a hearing-loss mutation R15G at 2.3-, 2.5-, and 2.6-Å resolutions, respectively. Compared with available structures of GJICh in open conformation, Cx31.3 hemichannel shows substantial structural changes of highly conserved regions in the connexin family, including opening of calcium ion-binding tunnels, reorganization of salt-bridge networks, exposure of lipid-binding sites, and collocation of amino-terminal helices at the cytoplasmic entrance. We also found that the hemichannel has a pore with a diameter of ~8 Å and selectively transports chloride ions. Our study provides structural insights into the permeant selectivity of Cx31.3 hemichannel. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0826.map.gz emd_0826.map.gz | 13.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0826-v30.xml emd-0826-v30.xml emd-0826.xml emd-0826.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0826_fsc.xml emd_0826_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0826.png emd_0826.png | 157.9 KB | ||
| Filedesc metadata |  emd-0826.cif.gz emd-0826.cif.gz | 5.4 KB | ||
| その他 |  emd_0826_half_map_1.map.gz emd_0826_half_map_1.map.gz emd_0826_half_map_2.map.gz emd_0826_half_map_2.map.gz | 96.9 MB 96.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0826 http://ftp.pdbj.org/pub/emdb/structures/EMD-0826 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0826 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0826 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0826.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0826.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocess_masked map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.673 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half map 2
| ファイル | emd_0826_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
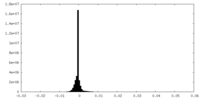
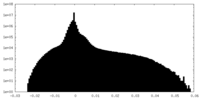
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_0826_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
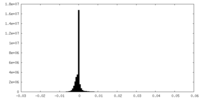
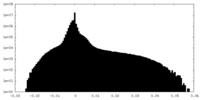
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human Cx31.3/GJC3 connexin hemichannel in the presence of calcium
| 全体 | 名称: Human Cx31.3/GJC3 connexin hemichannel in the presence of calcium |
|---|---|
| 要素 |
|
-超分子 #1: Human Cx31.3/GJC3 connexin hemichannel in the presence of calcium
| 超分子 | 名称: Human Cx31.3/GJC3 connexin hemichannel in the presence of calcium タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Gap junction gamma-3 protein
| 分子 | 名称: Gap junction gamma-3 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 31.339365 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MCGRFLRRLL AEESRRSTPV GRLLLPVLLG FRLVLLAASG PGVYGDEQSE FVCHTQQPGC KAACFDAFHP LSPLRFWVFQ VILVAVPSA LYMGFTLYHV IWHWELSGKG KEEETLIQGR EGNTDVPGAG SLRLLWAYVA QLGARLVLEG AALGLQYHLY G FQMPSSFA ...文字列: MCGRFLRRLL AEESRRSTPV GRLLLPVLLG FRLVLLAASG PGVYGDEQSE FVCHTQQPGC KAACFDAFHP LSPLRFWVFQ VILVAVPSA LYMGFTLYHV IWHWELSGKG KEEETLIQGR EGNTDVPGAG SLRLLWAYVA QLGARLVLEG AALGLQYHLY G FQMPSSFA CRREPCLGSI TCNLSRPSEK TIFLKTMFGV SGFCLLFTFL ELVLLGLGRW WRTWKHKSSS SKYFLTSEST RR HKKATDS LPVVETKEQF QEAVPGRSLA QEKQRPVGPR DA UniProtKB: Gap junction gamma-3 protein |
-分子 #2: Lauryl Maltose Neopentyl Glycol
| 分子 | 名称: Lauryl Maltose Neopentyl Glycol / タイプ: ligand / ID: 2 / コピー数: 6 / 式: LMN |
|---|---|
| 分子量 | 理論値: 1.005188 KDa |
| Chemical component information |  ChemComp-AV0: |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 6 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 174 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)