+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0789 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negatively stained reconstruction of a rubisco activase | |||||||||
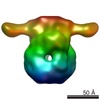
 マップデータ マップデータ | Negative stained 3D map of the rubisco activase | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Acidithiobacillus ferrooxidans (バクテリア) Acidithiobacillus ferrooxidans (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Tsai Y / Liu D / Bhushan S / Mueller-Cajar O | |||||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2020 ジャーナル: Proc Natl Acad Sci U S A / 年: 2020タイトル: Insights into the mechanism and regulation of the CbbQO-type Rubisco activase, a MoxR AAA+ ATPase. 著者: Yi-Chin Candace Tsai / Fuzhou Ye / Lynette Liew / Di Liu / Shashi Bhushan / Yong-Gui Gao / Oliver Mueller-Cajar /  要旨: The vast majority of biological carbon dioxide fixation relies on the function of ribulose 1,5-bisphosphate carboxylase/oxygenase (Rubisco). In most cases the enzyme exhibits a tendency to become ...The vast majority of biological carbon dioxide fixation relies on the function of ribulose 1,5-bisphosphate carboxylase/oxygenase (Rubisco). In most cases the enzyme exhibits a tendency to become inhibited by its substrate RuBP and other sugar phosphates. The inhibition is counteracted by diverse molecular chaperones known as Rubisco activases (Rcas). In some chemoautotrophic bacteria, the CbbQO-type Rca Q2O2 repairs inhibited active sites of hexameric form II Rubisco. The 2.2-Å crystal structure of the MoxR AAA+ protein CbbQ2 from reveals the helix 2 insert (H2I) that is critical for Rca function and forms the axial pore of the CbbQ hexamer. Negative-stain electron microscopy shows that the essential CbbO adaptor protein binds to the conserved, concave side of the CbbQ2 hexamer. Site-directed mutagenesis supports a model in which adenosine 5'-triphosphate (ATP)-powered movements of the H2I are transmitted to CbbO via the concave residue L85. The basal ATPase activity of Q2O2 Rca is repressed but strongly stimulated by inhibited Rubisco. The characterization of multiple variants where this repression is released indicates that binding of inhibited Rubisco to the C-terminal CbbO VWA domain initiates a signal toward the CbbQ active site that is propagated via elements that include the CbbQ α4-β4 loop, pore loop 1, and the presensor 1-β hairpin (PS1-βH). Detailed mechanistic insights into the enzyme repair chaperones of the highly diverse CO fixation machinery of Proteobacteria will facilitate their successful implementation in synthetic biology ventures. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0789.map.gz emd_0789.map.gz | 364.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0789-v30.xml emd-0789-v30.xml emd-0789.xml emd-0789.xml | 12.1 KB 12.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0789.png emd_0789.png | 28.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0789 http://ftp.pdbj.org/pub/emdb/structures/EMD-0789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0789 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0789 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0789_validation.pdf.gz emd_0789_validation.pdf.gz | 78.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0789_full_validation.pdf.gz emd_0789_full_validation.pdf.gz | 77.5 KB | 表示 | |
| XML形式データ |  emd_0789_validation.xml.gz emd_0789_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0789 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0789 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0789 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0789 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0789.map.gz / 形式: CCP4 / 大きさ: 489.3 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0789.map.gz / 形式: CCP4 / 大きさ: 489.3 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stained 3D map of the rubisco activase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.19 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CBBQO type Rubisco activase
| 全体 | 名称: CBBQO type Rubisco activase |
|---|---|
| 要素 |
|
-超分子 #1: CBBQO type Rubisco activase
| 超分子 | 名称: CBBQO type Rubisco activase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Acidithiobacillus ferrooxidans (バクテリア) Acidithiobacillus ferrooxidans (バクテリア) |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 0.27 kDa/nm |
-分子 #1: Rubisco activase
| 分子 | 名称: Rubisco activase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO |
|---|---|
| 由来(天然) | 生物種:  Acidithiobacillus ferrooxidans (バクテリア) Acidithiobacillus ferrooxidans (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTATDSSILN QYLVGKEPFY QPQHDEVALF EAAYRKRLPV MVKGPTGCGK SRFVEFMAWR LGKPLVTVAC NEDMTAADLV GRWLLDKDGT RWQDGPLTVA ARYGAICYLD EIVEARQDTT VVIHPLTDHR RTLPLDKKGE LIRAHPDFQL VISYNPGYQS LMKDLKQSTK ...文字列: MTATDSSILN QYLVGKEPFY QPQHDEVALF EAAYRKRLPV MVKGPTGCGK SRFVEFMAWR LGKPLVTVAC NEDMTAADLV GRWLLDKDGT RWQDGPLTVA ARYGAICYLD EIVEARQDTT VVIHPLTDHR RTLPLDKKGE LIRAHPDFQL VISYNPGYQS LMKDLKQSTK QRFTGFEFDY PNAELEAGIL VQETGVAPSI AAQLVTVAAT ARRLKGHGLD EGISTRLLVY AAMLMDDGVA PRAACRMALV QPITDDADIR ATLEHAIDMT FA MTDHPRD ASLPSRLAAY RKQLDCRFPR VGEVFSDCIA KASARLGPAG VSAYVDAARA LCKLGRGEEP VLIFLEEWPD VGAALGDGTL EMVMQMVQFM QRTPNGNAIG GFLQSLAPVS RALLSREQLG HYLNTLRDMM ERTTGSIHGH HQTHPSPGLP ELLRQAPTLL QSLTVDGLRN WVDYGVRNYL HHPERQKDFF SLQSADSRAV LQRERHGTLL ADVERKLDLY LRGLWQDSEV LVPYSTAFAT LRTPQPYYDA LGMRLPDVLD DLPGIGALDH YRAILAHMVG HRRWSTPQIA DNWSPFQRLA VEFFEDARID TLLIRTYPGL RTLFLALHPK PGEDACDPET TSCLRHRLAM LSRALLDPEH GYRNPVLHDF VDRFHGELAG GNADTATMAR LALDYVTTTR RQSDQFAKVH FADTEISYRD DNRGLWRFIE SGDEEEAFDA EQRKAPDLET QGLPPRHYPE WDYQSQSYRP DWVSLYEGLH ASAPAATIDQ LLLKHAALAK HLKRLLDLLK PQDKVRIRYQ EEGSELDLDV ALRSWIDFKS GSTPDPRINM SHRTAGRDIA VTLLLDLSES LNESVKTGGG DGQTVLQLSQ EAVSLLAWSI EQLGDPLAIA GFNSNTRHEV RYQHIKGFSE PWGDVVKGRL AALQAGYSTR MGAAMRHAGH YLATRKADKK LMLVLTDGRP SDVDVQDDRL LIEDARQAVN ELDRDGIFTY CISLDPHADA YVADIFGRQY TVIDHIARLP EKLPELFIAL TR |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 20 mM Tris-HCl pH8.0, 50 mM NaCl |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate 詳細: The sample was applied to a carbon-coated TEM grid and stained with 2% (w/v) uranyl acetate |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 6.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 詳細 | Monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 撮影したグリッド数: 1 / 実像数: 60 / 平均露光時間: 1.0 sec. / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 最大 デフォーカス(補正後): 1.5 µm / 最小 デフォーカス(補正後): 1.25 µm / 倍率(補正後): 49000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 6.3 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 1.25 µm |
| 試料ステージ | 試料ホルダーモデル: GATAN 910 MULTI-SPECIMEN SINGLE TILT CRYO TRANSFER HOLDER |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera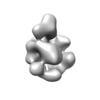






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















