+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0123 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the hub of the 36 triskelia D6 barrel clathrin coat complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
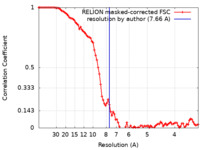
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.66 Å | |||||||||
 データ登録者 データ登録者 | Morris KL / Smith CJ | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2019 ジャーナル: Nat Struct Mol Biol / 年: 2019タイトル: Cryo-EM of multiple cage architectures reveals a universal mode of clathrin self-assembly. 著者: Kyle L Morris / Joseph R Jones / Mary Halebian / Shenping Wu / Michael Baker / Jean-Paul Armache / Amaurys Avila Ibarra / Richard B Sessions / Alexander D Cameron / Yifan Cheng / Corinne J Smith /   要旨: Clathrin forms diverse lattice and cage structures that change size and shape rapidly in response to the needs of eukaryotic cells during clathrin-mediated endocytosis and intracellular trafficking. ...Clathrin forms diverse lattice and cage structures that change size and shape rapidly in response to the needs of eukaryotic cells during clathrin-mediated endocytosis and intracellular trafficking. We present the cryo-EM structure and molecular model of assembled porcine clathrin, providing insights into interactions that stabilize key elements of the clathrin lattice, namely, between adjacent heavy chains, at the light chain-heavy chain interface and within the trimerization domain. Furthermore, we report cryo-EM maps for five different clathrin cage architectures. Fitting structural models to three of these maps shows that their assembly requires only a limited range of triskelion leg conformations, yet inherent flexibility is required to maintain contacts. Analysis of the protein-protein interfaces shows remarkable conservation of contact sites despite architectural variation. These data reveal a universal mode of clathrin assembly that allows variable cage architecture and adaptation of coated vesicle size and shape during clathrin-mediated vesicular trafficking or endocytosis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0123.map.gz emd_0123.map.gz | 8.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0123-v30.xml emd-0123-v30.xml emd-0123.xml emd-0123.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0123_fsc.xml emd_0123_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0123.png emd_0123.png | 105.9 KB | ||
| マスクデータ |  emd_0123_msk_1.map emd_0123_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0123_additional.map.gz emd_0123_additional.map.gz emd_0123_additional_1.map.gz emd_0123_additional_1.map.gz emd_0123_half_map_1.map.gz emd_0123_half_map_1.map.gz emd_0123_half_map_2.map.gz emd_0123_half_map_2.map.gz | 8.1 MB 8.1 MB 14.9 MB 14.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0123 http://ftp.pdbj.org/pub/emdb/structures/EMD-0123 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0123 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0123 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0123_validation.pdf.gz emd_0123_validation.pdf.gz | 321.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0123_full_validation.pdf.gz emd_0123_full_validation.pdf.gz | 320.9 KB | 表示 | |
| XML形式データ |  emd_0123_validation.xml.gz emd_0123_validation.xml.gz | 14.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0123 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0123 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0123 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0123 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  0114C  0115C  0116C  0118C  0120C  0121C  0122C  0124C  0125C  0126C  6sctC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10296 (タイトル: Single particle cryo-EM dataset of the triskelion hub subparticle extraction from clathrin cages EMPIAR-10296 (タイトル: Single particle cryo-EM dataset of the triskelion hub subparticle extraction from clathrin cagesData size: 88.4 Data #1: Hub subparticles of the 28 mini coat [picked particles - multiframe - unprocessed] Data #2: Hub subparticles of the 32 sweet potato [picked particles - multiframe - unprocessed] Data #3: Hub subparticles of the 36 D6 barrel [picked particles - multiframe - unprocessed] Data #4: Hub subparticles of the 36 tennis ball [picked particles - multiframe - unprocessed] Data #5: Hub subparticles of the 37 big apple [picked particles - multiframe - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0123.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0123.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.705 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
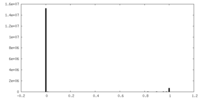
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0123_msk_1.map emd_0123_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
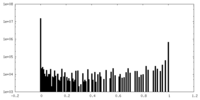
| 密度ヒストグラム |
-追加マップ: Post processed map from resolution measurement.
| ファイル | emd_0123_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post processed map from resolution measurement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
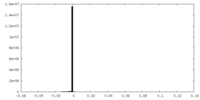
| 密度ヒストグラム |
-追加マップ: Post processed map from resolution measurement.
| ファイル | emd_0123_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post processed map from resolution measurement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
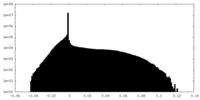
| 密度ヒストグラム |
-ハーフマップ: Soft spherical masked (diameter 320A) applied to remove...
| ファイル | emd_0123_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Soft spherical masked (diameter 320A) applied to remove artifactual density prior to resolution measurement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Soft spherical masked (diameter 320A) applied to remove...
| ファイル | emd_0123_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Soft spherical masked (diameter 320A) applied to remove artifactual density prior to resolution measurement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Assembly of clathrin heavy and light chain into coat complexes
| 全体 | 名称: Assembly of clathrin heavy and light chain into coat complexes |
|---|---|
| 要素 |
|
-超分子 #1: Assembly of clathrin heavy and light chain into coat complexes
| 超分子 | 名称: Assembly of clathrin heavy and light chain into coat complexes タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 540 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.32 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.4 構成要素:
| |||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: HOMEMADE PLUNGER 詳細: 3 uL applied to a grid at room temperature and humidity. 3 second hand blot and plunge.. | |||||||||||||||
| 詳細 | Clathrin coat complexes, end point assembly exhibiting architectural heterogeneity |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均露光時間: 3.0 sec. / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 82111 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.0032 µm / 最小 デフォーカス(公称値): 0.0014 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT 当てはまり具合の基準: Cross-correlation coefficient |
|---|
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)