+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 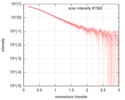 |
|---|---|
 試料 試料 | Thymine dioxygenase J-containing DNA binding domain in complex with J-23-DNA (JDBD:J-23-DNA)
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thymine dioxygenase / thymine dioxygenase activity / base J metabolic process / DNA binding / metal ion binding / nucleus 類似検索 - 分子機能 |
| 生物種 |  Leishmania tarentolae (真核生物) Leishmania tarentolae (真核生物) |
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2019 ジャーナル: J Biol Chem / 年: 2019タイトル: The domain architecture of the protozoan protein J-DNA-binding protein 1 suggests synergy between base J DNA binding and thymidine hydroxylase activity. 著者: Athanassios Adamopoulos / Tatjana Heidebrecht / Jeroen Roosendaal / Wouter G Touw / Isabelle Q Phan / Jos Beijnen / Anastassis Perrakis /   要旨: J-DNA-binding protein 1 (JBP1) contributes to the biosynthesis and maintenance of base J (β-d-glucosyl-hydroxymethyluracil), an epigenetic modification of thymidine (T) confined to pathogenic ...J-DNA-binding protein 1 (JBP1) contributes to the biosynthesis and maintenance of base J (β-d-glucosyl-hydroxymethyluracil), an epigenetic modification of thymidine (T) confined to pathogenic protozoa such as and JBP1 has two known functional domains: an N-terminal T hydroxylase (TH) homologous to the 5-methylcytosine hydroxylase domain in TET proteins and a J-DNA-binding domain (JDBD) that resides in the middle of JBP1. Here, we show that removing JDBD from JBP1 results in a soluble protein (Δ-JDBD) with the N- and C-terminal regions tightly associated together in a well-ordered structure. We found that this Δ-JDBD domain retains TH activity but displays a 15-fold lower apparent rate of hydroxylation compared with JBP1. Small-angle X-ray scattering (SAXS) experiments on JBP1 and JDBD in the presence or absence of J-DNA and on Δ-JDBD enabled us to generate low-resolution three-dimensional models. We conclude that Δ-JDBD, and not the N-terminal region of JBP1 alone, is a distinct folding unit. Our SAXS-based model supports the notion that binding of JDBD specifically to J-DNA can facilitate T hydroxylation 12-14 bp downstream on the complementary strand of the J-recognition site. We postulate that insertion of the JDBD module into the Δ-JDBD scaffold during evolution provided a mechanism that synergized J recognition and T hydroxylation, ensuring inheritance of base J in specific sequence patterns following DNA replication in kinetoplastid parasites. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Thymine dioxygenase J-containing DNA binding domain in complex with J-23-DNA (JDBD:J-23-DNA) Entity id: 898 / 899 |
|---|---|
| バッファ | 名称: 20 mM HEPES, 200 mM NaCl, 1 mM TCEP / pH: 7.5 |
| 要素 #898 | 名称: JDBD / タイプ: protein / 記述: J-DNA binding domain / 分子量: 21.244 / 分子数: 1 / 由来: Leishmania tarentolae / 参照: UniProt: Q9U6M1 配列: GPGPLSRLGG FSETNLMVST AVEKKKYLDS EFLLHCISAQ LLDMWKQARA RWLELVGKEW AHMLALNPER KDFLWKNQSE MNSAFFDLCE VGKQVMLGLL GKEVALPKEE QAFWIMYAVH LSAACAEELH MPEVAMSLRK LNVKLKDFNF GGTRYFKDMP PEEKKRRMER KQRIEEARRH GMP |
| 要素 #899 | 名称: J-23-DNA / タイプ: DNA / 記述: J-DNA (23mer) / 分子量: 14.225 / 分子数: 1 配列: TCGATTTGTT CATAGACTAA TACAGCTAAA CAAGTATCTG ATTATG |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 2.872 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 2.872 mm | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M / タイプ: Dectris / Pixsize x: 172 mm | ||||||||||||||||||||||||||||||
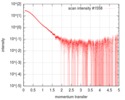
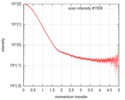
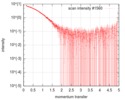
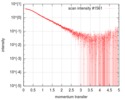
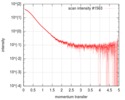
| スキャン | 測定日: 2016年2月21日 / セル温度: 4 °C / 照射時間: 1 sec. / フレーム数: 21 / 単位: 1/nm /
| ||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー



 SASDGT2
SASDGT2