+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 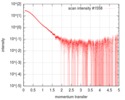 |
|---|---|
 試料 試料 | Thymine dioxygenase full length J-DNA binding protein (JBP1)
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thymine dioxygenase / thymine dioxygenase activity / base J metabolic process / DNA binding / metal ion binding / nucleus 類似検索 - 分子機能 |
| 生物種 |  Leishmania tarentolae (真核生物) Leishmania tarentolae (真核生物) |
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2019 ジャーナル: J Biol Chem / 年: 2019タイトル: The domain architecture of the protozoan protein J-DNA-binding protein 1 suggests synergy between base J DNA binding and thymidine hydroxylase activity. 著者: Athanassios Adamopoulos / Tatjana Heidebrecht / Jeroen Roosendaal / Wouter G Touw / Isabelle Q Phan / Jos Beijnen / Anastassis Perrakis /   要旨: J-DNA-binding protein 1 (JBP1) contributes to the biosynthesis and maintenance of base J (β-d-glucosyl-hydroxymethyluracil), an epigenetic modification of thymidine (T) confined to pathogenic ...J-DNA-binding protein 1 (JBP1) contributes to the biosynthesis and maintenance of base J (β-d-glucosyl-hydroxymethyluracil), an epigenetic modification of thymidine (T) confined to pathogenic protozoa such as and JBP1 has two known functional domains: an N-terminal T hydroxylase (TH) homologous to the 5-methylcytosine hydroxylase domain in TET proteins and a J-DNA-binding domain (JDBD) that resides in the middle of JBP1. Here, we show that removing JDBD from JBP1 results in a soluble protein (Δ-JDBD) with the N- and C-terminal regions tightly associated together in a well-ordered structure. We found that this Δ-JDBD domain retains TH activity but displays a 15-fold lower apparent rate of hydroxylation compared with JBP1. Small-angle X-ray scattering (SAXS) experiments on JBP1 and JDBD in the presence or absence of J-DNA and on Δ-JDBD enabled us to generate low-resolution three-dimensional models. We conclude that Δ-JDBD, and not the N-terminal region of JBP1 alone, is a distinct folding unit. Our SAXS-based model supports the notion that binding of JDBD specifically to J-DNA can facilitate T hydroxylation 12-14 bp downstream on the complementary strand of the J-recognition site. We postulate that insertion of the JDBD module into the Δ-JDBD scaffold during evolution provided a mechanism that synergized J recognition and T hydroxylation, ensuring inheritance of base J in specific sequence patterns following DNA replication in kinetoplastid parasites. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Thymine dioxygenase full length J-DNA binding protein (JBP1) 試料濃度: 8.7 mg/ml |
|---|---|
| バッファ | 名称: 20 mM HEPES, 200 mM NaCl, 1 mM TCEP / pH: 7.5 |
| 要素 #894 | 名称: JBP1 / タイプ: protein / 記述: Thymine dioxygenase JBP1 / 分子量: 93.482 / 分子数: 1 / 由来: Leishmania tarentolae / 参照: UniProt: Q9U6M1 配列: GPGEPDSKKV KLDIFNFPTT RETRTPEEVA ESYAEAVKSH PFYDNVHSVV DFYDSGTIKD GRGQIIGVVL REALPKYAAS MASELLTSAA VRTSLRSMMF GGEPPLSGIA GYFDYRGSPV ELKSRKTSFT YEHEAAWPAV FPVVDYVSEI YRHVAPERWK AQNDAIPDLV ...配列: GPGEPDSKKV KLDIFNFPTT RETRTPEEVA ESYAEAVKSH PFYDNVHSVV DFYDSGTIKD GRGQIIGVVL REALPKYAAS MASELLTSAA VRTSLRSMMF GGEPPLSGIA GYFDYRGSPV ELKSRKTSFT YEHEAAWPAV FPVVDYVSEI YRHVAPERWK AQNDAIPDLV RIHGTPFSTL TINSRFRTAS HTDVGDFDAG YSCIACLDGQ FKGLALSFDD FGINVLLQPR DVMIFDSHHF HSNTEVELSF SGEDWKRLTC VFYYRAALGE PASYAEYQRR LEKSKTDTRF TPVVHHVRVK ENGTSVNRPS PVYPISQSPF WVPMVAHCLQ HCASAAQCVH EAMTADGSRL AEMMFGESLS TSDGIPLRGE DEKVKANGDS TPRPLSRLGG FSETNLMVST AVEKKKYLDS EFLLHCISAQ LLDMWKQARA RWLELVGKEW AHMLALNPER KDFLWKNQSE MNSAFFDLCE VGKQVMLGLL GKEVALPKEE QAFWIMYAVH LSAACAEELH MPEVAMSLRK LNVKLKDFNF GGTRYFKDMP PEEKKRRMER KQRIEEARRH GMPSGSHEKR ANWLTNDSFD YQTEDCVIDY AQHKWVLPAL HAKEVTKTVR TGELPTTERV VRVLVVIPDP QSKLENVDCK LEVPDMVGSS SEWERLMSSP AVHRVLSAAQ RNLQLPDSVT HGNVQTHFAF HSTLPTDIYD FVVLQHVLSR IPDDAQASAY IRRAAALCSG CLFVVETDVQ CRQYYTLKYS IRCSYDTVAP LFFQQLHRVC YGTKTARVRT KGELESLIPT VCCARYKLQG SPLNTTVHVV SPFPSCEVQN LSSALCDRA |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 2.872 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 2.872 mm | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M / タイプ: Dectris / Pixsize x: 172 mm | ||||||||||||||||||||||||||||||
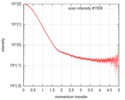
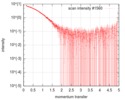
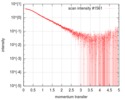
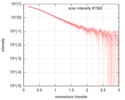
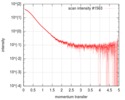
| スキャン | 測定日: 2016年2月21日 / セル温度: 4 °C / 照射時間: 1 sec. / フレーム数: 35 / 単位: 1/nm /
| ||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー



 SASDGP2
SASDGP2