+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 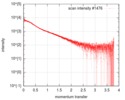 |
|---|---|
 試料 試料 | Delta subunit of RNA polymerase, RNAP (B. subtilis), 10mM NaCl
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA-directed RNA polymerase complex / DNA-directed RNA polymerase activity / DNA-templated transcription / regulation of DNA-templated transcription 類似検索 - 分子機能 |
| 生物種 |  |
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2019 ジャーナル: J Am Chem Soc / 年: 2019タイトル: Quantitative Conformational Analysis of Functionally Important Electrostatic Interactions in the Intrinsically Disordered Region of Delta Subunit of Bacterial RNA Polymerase. 著者: Vojtěch Kubáň / Pavel Srb / Hana Štégnerová / Petr Padrta / Milan Zachrdla / Zuzana Jaseňáková / Hana Šanderová / Dragana Vítovská / Libor Krásný / Tomáš Koval' / Jan ...著者: Vojtěch Kubáň / Pavel Srb / Hana Štégnerová / Petr Padrta / Milan Zachrdla / Zuzana Jaseňáková / Hana Šanderová / Dragana Vítovská / Libor Krásný / Tomáš Koval' / Jan Dohnálek / Joanna Ziemska-Legiecka / Marcin Grynberg / Patryk Jarnot / Aleksandra Gruca / Malene Ringkjøbing Jensen / Martin Blackledge / Lukáš Žídek /    要旨: Electrostatic interactions play important roles in the functional mechanisms exploited by intrinsically disordered proteins (IDPs). The atomic resolution description of long-range and local ...Electrostatic interactions play important roles in the functional mechanisms exploited by intrinsically disordered proteins (IDPs). The atomic resolution description of long-range and local structural propensities that can both be crucial for the function of highly charged IDPs presents significant experimental challenges. Here, we investigate the conformational behavior of the δ subunit of RNA polymerase from whose unfolded domain is highly charged, with 7 positively charged amino acids followed by 51 acidic amino acids. Using a specifically designed analytical strategy, we identify transient contacts between the two regions using a combination of NMR paramagnetic relaxation enhancements, residual dipolar couplings (RDCs), chemical shifts, and small-angle scattering. This strategy allows the resolution of long-range and local ensemble averaged structural contributions to the experimental RDCs, and reveals that the negatively charged segment folds back onto the positively charged strand, compacting the conformational sampling of the protein while remaining highly flexible in solution. Mutation of the positively charged region abrogates the long-range contact, leaving the disordered domain in an extended conformation, possibly due to local repulsion of like-charges along the chain. Remarkably, in vitro studies show that this mutation also has a significant effect on transcription activity, and results in diminished cell fitness of the mutated bacteria in vivo. This study highlights the importance of accurately describing electrostatic interactions for understanding the functional mechanisms of IDPs. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Delta subunit of RNA polymerase, RNAP (B. subtilis), 10mM NaCl 試料濃度: 0.90-3.60 |
|---|---|
| バッファ | 名称: 20 mM Phosphate buffer, 10 mM NaCl, 0.05% NaN3 / pH: 6.6 |
| 要素 #1736 | 名称: RpoE / タイプ: protein / 記述: DNA-directed RNA polymerase subunit delta / 分子量: 20.399 / 分子数: 1 / 由来: Bacillus subtilis (strain 168) / 参照: UniProt: P12464 配列: MGIKQYSQEE LKEMALVEIA HELFEEHKKP VPFQELLNEI ASLLGVKKEE LGDRIAQFYT DLNIDGRFLA LSDQTWGLRS WYPYDQLDEE TQPTVKAKKK KAKKAVEEDL DLDEFEEIDE DDLDLDEVEE ELDLEADDFD EEDLDEDDDD LEIEEDIIDE DDEDYDDEEE EIK |
-実験情報
| ビーム | 設備名称: PETRA III EMBL P12 / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3 mm / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3 mm | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | |||||||||||||||||||||||||||
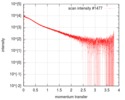
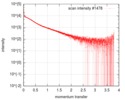
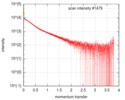
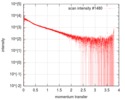
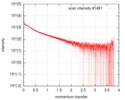
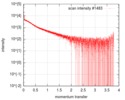
| スキャン | 測定日: 2016年10月3日 / 保管温度: 20 °C / セル温度: 20 °C / 照射時間: 0.05 sec. / フレーム数: 20 / 単位: 1/nm /
| |||||||||||||||||||||||||||
| 結果 | カーブのタイプ: extrapolated コメント: There is a concentration effect for the delta subunit of RNA polymerase in a low salt concentration buffer.
|
 ムービー
ムービー コントローラー
コントローラー



 SASDF58
SASDF58