+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | Unlabeled nuclear pore complex protein Nup98-Nup96 (N98) without denaturant
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nuclear pore complex assembly / telomere tethering at nuclear periphery / nuclear pore outer ring / nuclear pore organization / nuclear pore cytoplasmic filaments / post-transcriptional tethering of RNA polymerase II gene DNA at nuclear periphery / Nuclear Pore Complex (NPC) Disassembly / nuclear inclusion body / nuclear pore nuclear basket / Transport of Ribonucleoproteins into the Host Nucleus ...nuclear pore complex assembly / telomere tethering at nuclear periphery / nuclear pore outer ring / nuclear pore organization / nuclear pore cytoplasmic filaments / post-transcriptional tethering of RNA polymerase II gene DNA at nuclear periphery / Nuclear Pore Complex (NPC) Disassembly / nuclear inclusion body / nuclear pore nuclear basket / Transport of Ribonucleoproteins into the Host Nucleus / Regulation of Glucokinase by Glucokinase Regulatory Protein / Defective TPR may confer susceptibility towards thyroid papillary carcinoma (TPC) / Transport of the SLBP independent Mature mRNA / NS1 Mediated Effects on Host Pathways / Transport of the SLBP Dependant Mature mRNA / SUMOylation of SUMOylation proteins / structural constituent of nuclear pore / nuclear localization sequence binding / Transport of Mature mRNA Derived from an Intronless Transcript / positive regulation of mRNA splicing, via spliceosome / Rev-mediated nuclear export of HIV RNA / Nuclear import of Rev protein / SUMOylation of RNA binding proteins / NEP/NS2 Interacts with the Cellular Export Machinery / RNA export from nucleus / Transport of Mature mRNA derived from an Intron-Containing Transcript / tRNA processing in the nucleus / Postmitotic nuclear pore complex (NPC) reformation / nucleocytoplasmic transport / Viral Messenger RNA Synthesis / SUMOylation of ubiquitinylation proteins / Vpr-mediated nuclear import of PICs / SUMOylation of DNA replication proteins / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / Regulation of HSF1-mediated heat shock response / mRNA transport / nuclear pore / SUMOylation of DNA damage response and repair proteins / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / Mitotic Prometaphase / serine-type peptidase activity / EML4 and NUDC in mitotic spindle formation / nuclear periphery / Resolution of Sister Chromatid Cohesion / SUMOylation of chromatin organization proteins / HCMV Late Events / promoter-specific chromatin binding / Transcriptional regulation by small RNAs / RHO GTPases Activate Formins / ISG15 antiviral mechanism / molecular condensate scaffold activity / Separation of Sister Chromatids / HCMV Early Events / protein import into nucleus / nuclear envelope / snRNP Assembly / nuclear membrane / transcription coactivator activity / nuclear body / ribonucleoprotein complex / mRNA binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / proteolysis / RNA binding / nucleoplasm / cytosol 類似検索 - 分子機能 |
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2017 ジャーナル: Proc Natl Acad Sci U S A / 年: 2017タイトル: Decoupling of size and shape fluctuations in heteropolymeric sequences reconciles discrepancies in SAXS vs. FRET measurements. 著者: Gustavo Fuertes / Niccolò Banterle / Kiersten M Ruff / Aritra Chowdhury / Davide Mercadante / Christine Koehler / Michael Kachala / Gemma Estrada Girona / Sigrid Milles / Ankur Mishra / ...著者: Gustavo Fuertes / Niccolò Banterle / Kiersten M Ruff / Aritra Chowdhury / Davide Mercadante / Christine Koehler / Michael Kachala / Gemma Estrada Girona / Sigrid Milles / Ankur Mishra / Patrick R Onck / Frauke Gräter / Santiago Esteban-Martín / Rohit V Pappu / Dmitri I Svergun / Edward A Lemke /     要旨: Unfolded states of proteins and native states of intrinsically disordered proteins (IDPs) populate heterogeneous conformational ensembles in solution. The average sizes of these heterogeneous ...Unfolded states of proteins and native states of intrinsically disordered proteins (IDPs) populate heterogeneous conformational ensembles in solution. The average sizes of these heterogeneous systems, quantified by the radius of gyration ( ), can be measured by small-angle X-ray scattering (SAXS). Another parameter, the mean dye-to-dye distance ( ) for proteins with fluorescently labeled termini, can be estimated using single-molecule Förster resonance energy transfer (smFRET). A number of studies have reported inconsistencies in inferences drawn from the two sets of measurements for the dimensions of unfolded proteins and IDPs in the absence of chemical denaturants. These differences are typically attributed to the influence of fluorescent labels used in smFRET and to the impact of high concentrations and averaging features of SAXS. By measuring the dimensions of a collection of labeled and unlabeled polypeptides using smFRET and SAXS, we directly assessed the contributions of dyes to the experimental values and For chemically denatured proteins we obtain mutual consistency in our inferences based on and , whereas for IDPs under native conditions, we find substantial deviations. Using computations, we show that discrepant inferences are neither due to methodological shortcomings of specific measurements nor due to artifacts of dyes. Instead, our analysis suggests that chemical heterogeneity in heteropolymeric systems leads to a decoupling between and that is amplified in the absence of denaturants. Therefore, joint assessments of and combined with measurements of polymer shapes should provide a consistent and complete picture of the underlying ensembles. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-Data source
| SASBDBのページ |  SASDEB3 SASDEB3 |
|---|
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- 外部リンク
外部リンク
| 「今月の分子」の関連する項目 |
|---|
-モデル
- 試料
試料
 試料 試料 | 名称: Unlabeled nuclear pore complex protein Nup98-Nup96 (N98) without denaturant 試料濃度: 1.00-10.00 |
|---|---|
| バッファ | 名称: PBS, 10 mM DTT / pH: 7.4 |
| 要素 #1248 | 名称: N98 / タイプ: protein / 記述: Nuclear pore complex protein Nup98-Nup96 / 分子量: 14.763 / 分子数: 1 / 由来: Homo sapiens / 参照: UniProt: P52948 配列: GCFNKSFGTP FGGGTGGFGT TSTFGQNTGF GTTSGGAFGT SAFGSSNNTG GLFGNSQTKP GGLFGTSSFS QPATSTSTGF GFGTSTGTAN TLFGTASTGT SLFSSQNNAF AQNKPTGFGN FGTSTSSGGL FGTTNTTSNP FGSTSGSLFG PUA |
-実験情報
| ビーム | 設備名称: PETRA III EMBL P12 / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 3 mm / 線源: X-ray synchrotron / 波長: 0.1 Å / スペクトロメータ・検出器間距離: 3 mm | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | |||||||||||||||||||||||||||||||||
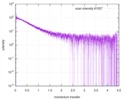
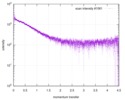
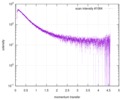
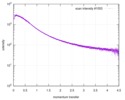
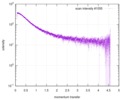
| スキャン | 測定日: 2015年6月24日 / セル温度: 23 °C / 照射時間: 0.05 sec. / フレーム数: 20 / 単位: 1/nm /
| |||||||||||||||||||||||||||||||||
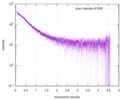
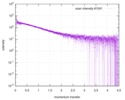
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||
| 結果 | コメント: The protein contains a penultimate non-canonical amino acid p-acetylphenylalanine (207 Da) that is represented as U (selenocysteine, 168 Da) in the amino acid sequence for the entry. ...コメント: The protein contains a penultimate non-canonical amino acid p-acetylphenylalanine (207 Da) that is represented as U (selenocysteine, 168 Da) in the amino acid sequence for the entry. Therefore, the calculated MW from sequence (MW(expected)) must be adjusted accordingly (ca. 40 Da).
|
 ムービー
ムービー コントローラー
コントローラー