+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | p-hydroxyphenylacetate 3-hydroxylase, reductase component (mutant): 4 mg/ml of Y207A C1 in the absence of p-hydroxyphenylacetic acid (HPA)
|
| 生物種 |  Acinetobacter baumannii (バクテリア) Acinetobacter baumannii (バクテリア) |
 引用 引用 |  ジャーナル: Arch Biochem Biophys / 年: 2018 ジャーナル: Arch Biochem Biophys / 年: 2018タイトル: Crystal structure of the flavin reductase of Acinetobacter baumannii p-hydroxyphenylacetate 3-hydroxylase (HPAH) and identification of amino acid residues underlying its regulation by aromatic ligands. 著者: Anan Yuenyao / Nopphon Petchyam / Nuntaporn Kamonsutthipaijit / Pimchai Chaiyen / Danaya Pakotiprapha /  要旨: The first step in the degradation of p-hydroxyphenylacetic acid (HPA) is catalyzed by the two-component enzyme p-hydroxyphenylacetate 3-hydroxylase (HPAH). The two components of Acinetobacter ...The first step in the degradation of p-hydroxyphenylacetic acid (HPA) is catalyzed by the two-component enzyme p-hydroxyphenylacetate 3-hydroxylase (HPAH). The two components of Acinetobacter baumannii HPAH are known as C and C, respectively. C is a flavin reductase that uses NADH to generate reduced flavin mononucleotide (FMNH), which is used by C in the hydroxylation of HPA. Interestingly, although HPA is not directly involved in the reaction catalyzed by C, the presence of HPA dramatically increases the FMN reduction rate. Amino acid sequence analysis revealed that C contains two domains: an N-terminal flavin reductase domain, and a C-terminal MarR domain. Although MarR proteins typically function as transcription regulators, the MarR domain of C was found to play an auto-inhibitory role. Here, we report a crystal structure of C and small-angle X-ray scattering (SAXS) studies that revealed that C undergoes a substantial conformational change in the presence of HPA, concomitant with the increase in the rate of flavin reduction. Amino acid residues that are important for HPA binding and regulation of C activity were identified by site-directed mutagenesis. Amino acid sequence similarity analysis revealed several as yet uncharacterized flavin reductases with N- or C-terminal fusions. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: p-hydroxyphenylacetate 3-hydroxylase, reductase component (mutant): 4 mg/ml of Y207A C1 in the absence of p-hydroxyphenylacetic acid (HPA) 試料濃度: 4 mg/ml |
|---|---|
| バッファ | 名称: 50 mM MOPS, 0.5 mM EDTA, 1 mM DTT, and 5% glycerol / pH: 7 |
| 要素 #985 | 名称: Y207A C1 reductase / タイプ: protein 記述: p-hydroxyphenylacetate 3-hydroxylase (HPAH), reductase component Y207A mutant 分子量: 35.32 / 分子数: 2 / 由来: Acinetobacter baumannii 配列: MNQLNTAIVE KEVIDPMAFR RALGNFATGV TIMTAQTSSG ERVGVTANSF NSVSLDPALV LWSIDKKSSS YRIFEEATHF GVNILSAAQI ELSNRFARRS EDKFANIEFD LGVGNIPLFK NCSAAFECER YNIVEGGDHW IIIGRVVKFH DHGRSPLLYH QGAYSAVLPH ...配列: MNQLNTAIVE KEVIDPMAFR RALGNFATGV TIMTAQTSSG ERVGVTANSF NSVSLDPALV LWSIDKKSSS YRIFEEATHF GVNILSAAQI ELSNRFARRS EDKFANIEFD LGVGNIPLFK NCSAAFECER YNIVEGGDHW IIIGRVVKFH DHGRSPLLYH QGAYSAVLPH PSLNMKSETA EGVFPGRLYD NMYYLLTQAV RAYQNDAQPK QLASGFRTSE ARLLLVLESK TASSKCDLQR EVAMPIREIE EATKILSEKG LLIDNGQHYE LTEQGNACAH MLYKIAESHQ EEVFAKYTVD ERKLFKNMLK DLIGI |
-実験情報
| ビーム | 設備名称: Synchrotron Light Research Institute (SLRI) BL1.3W 地域: Nakhon Ratchasima / 国: Thailand  / 線源: X-ray synchrotron / 波長: 0.13776 Å / スペクトロメータ・検出器間距離: 1.3323 mm / 線源: X-ray synchrotron / 波長: 0.13776 Å / スペクトロメータ・検出器間距離: 1.3323 mm | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Rayonix SX165 / タイプ: CCD / Pixsize x: 15 mm | ||||||||||||||||||||||||||||||
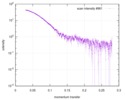
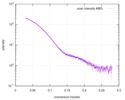
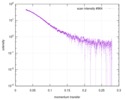
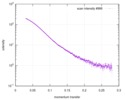
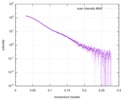
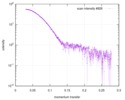
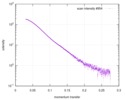
| スキャン | 測定日: 2017年3月7日 / 保管温度: 4 °C / セル温度: 16 °C / 照射時間: 600 sec. / フレーム数: 1 / 単位: 1/A /
| ||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| ||||||||||||||||||||||||||||||
| 結果 |
|
 ムービー
ムービー コントローラー
コントローラー 万見について
万見について



 SASDDK4
SASDDK4