+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  |
|---|---|
 試料 試料 | Candida antarctica lipase B - with guanidine-HCl unfolding series
|
| 機能・相同性 | : / triacylglycerol lipase / triacylglycerol lipase activity / lipid catabolic process / Alpha/Beta hydrolase fold / Lipase B 機能・相同性情報 機能・相同性情報 |
| 生物種 | Pseudozyma antarctica (Candida antarctica) |
 引用 引用 |  ジャーナル: Biophys J / 年: 2018 ジャーナル: Biophys J / 年: 2018タイトル: Machine Learning Methods for X-Ray Scattering Data Analysis from Biomacromolecular Solutions. 著者: Daniel Franke / Cy M Jeffries / Dmitri I Svergun /  要旨: Small-angle x-ray scattering (SAXS) of biological macromolecules in solutions is a widely employed method in structural biology. SAXS patterns include information about the overall shape and low- ...Small-angle x-ray scattering (SAXS) of biological macromolecules in solutions is a widely employed method in structural biology. SAXS patterns include information about the overall shape and low-resolution structure of dissolved particles. Here, we describe how to transform experimental SAXS patterns to feature vectors and how a simple k-nearest neighbor approach is able to retrieve information on overall particle shape and maximal diameter (D) as well as molecular mass directly from experimental scattering data. Based on this transformation, we develop a rapid multiclass shape-classification ranging from compact, extended, and flat categories to hollow and random-chain-like objects. This classification may be employed, e.g., as a decision block in automated data analysis pipelines. Further, we map protein structures from the Protein Data Bank into the classification space and, in a second step, use this mapping as a data source to obtain accurate estimates for the structural parameters (D, molecular mass) of the macromolecule under study based on the experimental scattering pattern alone, without inverse Fourier transform for D. All methods presented are implemented in a Fortran binary DATCLASS, part of the ATSAS data analysis suite, available on Linux, Mac, and Windows and free for academic use. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Candida antarctica lipase B - with guanidine-HCl unfolding series 試料濃度: 4.65 mg/ml |
|---|---|
| バッファ | 名称: 100 mM NaCl, 20 mM Na2HPO4 / pH: 6 |
| 要素 #968 | 名称: lipase B / タイプ: protein / 記述: Lipase B from Pseudozyma antarctica / 分子量: 33.022 / 由来: Pseudozyma antarctica (Candida antarctica) / 参照: UniProt: P41365 配列: LPSGSDPAFS QPKSVLDAGL TCQGASPSSV SKPILLVPGT GTTGPQSFDS NWIPLSTQLG YTPCWISPPP FMLNDTQVNT EYMVNAITAL YAGSGNNKLP VLTWSQGGLV AQWGLTFFPS IRSKVDRLMA FAPDYKGTVL AGPLDALAVS APSVWQQTTG SALTTALRNA ...配列: LPSGSDPAFS QPKSVLDAGL TCQGASPSSV SKPILLVPGT GTTGPQSFDS NWIPLSTQLG YTPCWISPPP FMLNDTQVNT EYMVNAITAL YAGSGNNKLP VLTWSQGGLV AQWGLTFFPS IRSKVDRLMA FAPDYKGTVL AGPLDALAVS APSVWQQTTG SALTTALRNA GGLTQIVPTT NLYSATDEIV QPQVSNSPLD SSYLFNGKNV QAQAVCGPLF VIDHAGSLTS QFSYVVGRSA LRSTTGQARS ADYGITDCNP LPANDLTPEQ KVAAAALLAP AAAAIVAGPK QNCEPDLMPY ARPFAVGKRT CSGIVTP |
-実験情報
| ビーム | 設備名称: PETRA III EMBL P12 / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3.1 mm / 線源: X-ray synchrotron / 波長: 0.124 Å / スペクトロメータ・検出器間距離: 3.1 mm | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | |||||||||||||||||||||
| スキャン | 測定日: 2013年7月29日 / 保管温度: 10 °C / セル温度: 10 °C / 照射時間: 0.03 sec. / フレーム数: 1 / 単位: 1/nm /
| |||||||||||||||||||||
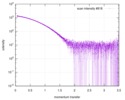
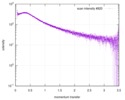
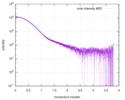
| 結果 | カーブのタイプ: single_conc コメント: In addition to the SAXS profile displayed above, a lipase B unfolding series spanning 0 M to 6 M guanidine hydrochloride is included in the full entry zip archive.
|
 ムービー
ムービー コントローラー
コントローラー



 SASDDJ3
SASDDJ3