+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8rb8 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the NADH:ferredoxin oxidoreductase RNF from Azotobacter vinelandii, purified with 2-ME/TCEP, NADH added | ||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN / NADH:ferredoxin oxidoreductase | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報トランスロカーゼ / electron transport chain / transmembrane transport / FMN binding / 4 iron, 4 sulfur cluster binding / electron transfer activity / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) | ||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.41 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Zhang, L. / Einsle, O. | ||||||||||||||||||
| 資金援助 | European Union,  ドイツ, 5件 ドイツ, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2024 ジャーナル: Nat Chem Biol / 年: 2024タイトル: Architecture of the RNF1 complex that drives biological nitrogen fixation. 著者: Lin Zhang / Oliver Einsle /  要旨: Biological nitrogen fixation requires substantial metabolic energy in form of ATP as well as low-potential electrons that must derive from central metabolism. During aerobic growth, the free-living ...Biological nitrogen fixation requires substantial metabolic energy in form of ATP as well as low-potential electrons that must derive from central metabolism. During aerobic growth, the free-living soil diazotroph Azotobacter vinelandii transfers electrons from the key metabolite NADH to the low-potential ferredoxin FdxA that serves as a direct electron donor to the dinitrogenase reductases. This process is mediated by the RNF complex that exploits the proton motive force over the cytoplasmic membrane to lower the midpoint potential of the transferred electron. Here we report the cryogenic electron microscopy structure of the nitrogenase-associated RNF complex of A. vinelandii, a seven-subunit membrane protein assembly that contains four flavin cofactors and six iron-sulfur centers. Its function requires the strict coupling of electron and proton transfer but also involves major conformational changes within the assembly that can be traced with a combination of electron microscopy and modeling. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8rb8.cif.gz 8rb8.cif.gz | 331.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8rb8.ent.gz pdb8rb8.ent.gz | 261.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8rb8.json.gz 8rb8.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8rb8_validation.pdf.gz 8rb8_validation.pdf.gz | 2.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8rb8_full_validation.pdf.gz 8rb8_full_validation.pdf.gz | 2.3 MB | 表示 | |
| XML形式データ |  8rb8_validation.xml.gz 8rb8_validation.xml.gz | 56.5 KB | 表示 | |
| CIF形式データ |  8rb8_validation.cif.gz 8rb8_validation.cif.gz | 82.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rb/8rb8 https://data.pdbj.org/pub/pdb/validation_reports/rb/8rb8 ftp://data.pdbj.org/pub/pdb/validation_reports/rb/8rb8 ftp://data.pdbj.org/pub/pdb/validation_reports/rb/8rb8 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  19028MC  8ahxC  8rb9C  8rbmC  8rbqC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Ion-translocating oxidoreductase complex subunit ... , 6種, 6分子 ACDEGB
| #1: タンパク質 | 分子量: 19942.131 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定)遺伝子: rnfA1, rnfA, Avin_50980 / 発現宿主:  Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA8, トランスロカーゼ Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA8, トランスロカーゼ |
|---|---|
| #2: タンパク質 | 分子量: 52234.195 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定)遺伝子: rnfC1, rnfC, Avin_50960 / 発現宿主:  Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA6, トランスロカーゼ Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA6, トランスロカーゼ |
| #3: タンパク質 | 分子量: 39189.016 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定)遺伝子: rnfD1, rnfD, Avin_50950 / 発現宿主:  Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA5, トランスロカーゼ Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA5, トランスロカーゼ |
| #4: タンパク質 | 分子量: 25576.328 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定)遺伝子: rnfE, Avin_50930 / 発現宿主:  Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: Q9F5Y1, トランスロカーゼ Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: Q9F5Y1, トランスロカーゼ |
| #5: タンパク質 | 分子量: 25932.709 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定)遺伝子: rnfG1, rnfG, Avin_50940 / 発現宿主:  Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA4, トランスロカーゼ Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA4, トランスロカーゼ |
| #7: タンパク質 | 分子量: 17704.572 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定)遺伝子: rnfB1, rnfB, Avin_50970 / 発現宿主:  Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA7, トランスロカーゼ Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: C1DMA7, トランスロカーゼ |
-タンパク質 / 糖 , 2種, 14分子 H

| #6: タンパク質 | 分子量: 9644.859 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定)遺伝子: rnfH, Avin_50920 / 発現宿主:  Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: Q9F5Y0 Azotobacter vinelandii DJ (窒素固定) / 参照: UniProt: Q9F5Y0 |
|---|---|
| #9: 糖 | ChemComp-LMT / |
-非ポリマー , 4種, 9分子 


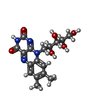



| #8: 化合物 | ChemComp-FES / | ||||
|---|---|---|---|---|---|
| #10: 化合物 | | #11: 化合物 | ChemComp-SF4 / #12: 化合物 | ChemComp-RBF / | |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Heptameric complex NADH:ferredoxin oxidoreductase RNF1 タイプ: COMPLEX / Entity ID: #1-#7 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.188 MDa / 実験値: NO |
| 由来(天然) | 生物種:  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) |
| 由来(組換発現) | 生物種:  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 37 e/Å2 フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) |
- 解析
解析
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 3次元再構成 | 解像度: 3.41 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 92067 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj
















