+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8q7h | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
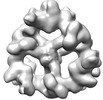
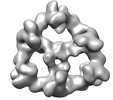
| タイトル | Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | LIGASE / Cullin-RING RBR E3 Ligase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / regulation of proteolysis / positive regulation of protein autoubiquitination / protein neddylation ...cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / regulation of proteolysis / positive regulation of protein autoubiquitination / protein neddylation / NEDD8 ligase activity / Cul5-RING ubiquitin ligase complex / negative regulation of response to oxidative stress / ubiquitin-ubiquitin ligase activity / Cul4A-RING E3 ubiquitin ligase complex / SCF ubiquitin ligase complex / Cul2-RING ubiquitin ligase complex / negative regulation of type I interferon production / Cul4B-RING E3 ubiquitin ligase complex / regulation of mitotic nuclear division / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / Cul3-RING ubiquitin ligase complex / Prolactin receptor signaling / TGF-beta receptor signaling activates SMADs / protein monoubiquitination / cullin family protein binding / anatomical structure morphogenesis / Nuclear events stimulated by ALK signaling in cancer / protein K48-linked ubiquitination / positive regulation of TORC1 signaling / T cell activation / regulation of cellular response to insulin stimulus / Regulation of BACH1 activity / post-translational protein modification / Degradation of DVL / Recognition of DNA damage by PCNA-containing replication complex / Degradation of GLI1 by the proteasome / Negative regulation of NOTCH4 signaling / cellular response to amino acid stimulus / Iron uptake and transport / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Vif-mediated degradation of APOBEC3G / Hedgehog 'on' state / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / DNA Damage Recognition in GG-NER / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / RING-type E3 ubiquitin transferase / Degradation of beta-catenin by the destruction complex / protein modification process / negative regulation of canonical Wnt signaling pathway / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / protein localization / Dual Incision in GG-NER / Evasion by RSV of host interferon responses / NOTCH1 Intracellular Domain Regulates Transcription / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex / Constitutive Signaling by NOTCH1 PEST Domain Mutants / Constitutive Signaling by NOTCH1 HD+PEST Domain Mutants / Regulation of expression of SLITs and ROBOs / Formation of Incision Complex in GG-NER / modification-dependent protein catabolic process / microtubule cytoskeleton organization / Interleukin-1 signaling / Orc1 removal from chromatin / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / Regulation of RAS by GAPs / protein polyubiquitination / positive regulation of protein catabolic process / protein tag activity / Regulation of RUNX2 expression and activity / UCH proteinases / cellular response to UV / KEAP1-NFE2L2 pathway / MAPK cascade / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / Cargo recognition for clathrin-mediated endocytosis / Neddylation / ubiquitin-dependent protein catabolic process / spermatogenesis / proteasome-mediated ubiquitin-dependent protein catabolic process / positive regulation of canonical NF-kappaB signal transduction / RNA polymerase II-specific DNA-binding transcription factor binding / Potential therapeutics for SARS / molecular adaptor activity / protein ubiquitination / DNA repair / ubiquitin protein ligase binding / DNA damage response / regulation of transcription by RNA polymerase II / proteolysis / zinc ion binding / extracellular exosome / nucleoplasm / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Hopf, L.V.M. / Horn-Ghetko, D. / Schulman, B.A. | |||||||||
| 資金援助 | European Union,  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Noncanonical assembly, neddylation and chimeric cullin-RING/RBR ubiquitylation by the 1.8 MDa CUL9 E3 ligase complex. 著者: Daniel Horn-Ghetko / Linus V M Hopf / Ishita Tripathi-Giesgen / Jiale Du / Sebastian Kostrhon / D Tung Vu / Viola Beier / Barbara Steigenberger / J Rajan Prabu / Luca Stier / Elias M Bruss / ...著者: Daniel Horn-Ghetko / Linus V M Hopf / Ishita Tripathi-Giesgen / Jiale Du / Sebastian Kostrhon / D Tung Vu / Viola Beier / Barbara Steigenberger / J Rajan Prabu / Luca Stier / Elias M Bruss / Matthias Mann / Yue Xiong / Brenda A Schulman /   要旨: Ubiquitin ligation is typically executed by hallmark E3 catalytic domains. Two such domains, 'cullin-RING' and 'RBR', are individually found in several hundred human E3 ligases, and collaborate with ...Ubiquitin ligation is typically executed by hallmark E3 catalytic domains. Two such domains, 'cullin-RING' and 'RBR', are individually found in several hundred human E3 ligases, and collaborate with E2 enzymes to catalyze ubiquitylation. However, the vertebrate-specific CUL9 complex with RBX1 (also called ROC1), of interest due to its tumor suppressive interaction with TP53, uniquely encompasses both cullin-RING and RBR domains. Here, cryo-EM, biochemistry and cellular assays elucidate a 1.8-MDa hexameric human CUL9-RBX1 assembly. Within one dimeric subcomplex, an E2-bound RBR domain is activated by neddylation of its own cullin domain and positioning from the adjacent CUL9-RBX1 in trans. Our data show CUL9 as unique among RBX1-bound cullins in dependence on the metazoan-specific UBE2F neddylation enzyme, while the RBR domain protects it from deneddylation. Substrates are recruited to various upstream domains, while ubiquitylation relies on both CUL9's neddylated cullin and RBR domains achieving self-assembled and chimeric cullin-RING/RBR E3 ligase activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8q7h.cif.gz 8q7h.cif.gz | 501.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8q7h.ent.gz pdb8q7h.ent.gz | 350.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8q7h.json.gz 8q7h.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8q7h_validation.pdf.gz 8q7h_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8q7h_full_validation.pdf.gz 8q7h_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  8q7h_validation.xml.gz 8q7h_validation.xml.gz | 83.7 KB | 表示 | |
| CIF形式データ |  8q7h_validation.cif.gz 8q7h_validation.cif.gz | 132.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/q7/8q7h https://data.pdbj.org/pub/pdb/validation_reports/q7/8q7h ftp://data.pdbj.org/pub/pdb/validation_reports/q7/8q7h ftp://data.pdbj.org/pub/pdb/validation_reports/q7/8q7h | HTTPS FTP |
-関連構造データ
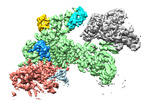
| 関連構造データ | 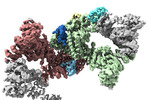 18216MC  8q7eC  8rhzC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 281686.062 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CUL9, H7AP1, KIAA0708, PARC / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CUL9, H7AP1, KIAA0708, PARC / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q8IWT3 Homo sapiens (ヒト) / 参照: UniProt: Q8IWT3#2: タンパク質 | 分子量: 12289.977 Da / 分子数: 2 / Mutation: p.M1_A4del / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: RBX1, RNF75, ROC1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: RBX1, RNF75, ROC1 / 発現宿主:  Homo sapiens (ヒト) Homo sapiens (ヒト)参照: UniProt: P62877, RING-type E3 ubiquitin transferase, cullin-RING-type E3 NEDD8 transferase #3: タンパク質 | | 分子量: 9086.562 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 参照: UniProt: Q15843 Homo sapiens (ヒト) / 参照: UniProt: Q15843#4: タンパク質 | | 分子量: 12868.854 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) Homo sapiens (ヒト)#5: 化合物 | ChemComp-ZN / 研究の焦点であるリガンドがあるか | Y | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Structure of CUL9-RBX1 ubiquitin E3 ligase complex in unneddylated and neddylated conformation - focused cullin dimer タイプ: COMPLEX / 詳細: Structure of dimeric subunit / Entity ID: #1-#4 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 緩衝液 | pH: 7.5 |
| 試料 | 濃度: 3 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2800 nm / 最小 デフォーカス(公称値): 700 nm |
| 撮影 | 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / バージョン: 1.17.1_3660: / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 4.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 71928 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj







