+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8pm2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | MEMBRANE PROTEIN / trace-amine associated receptor / TAAR / mTAAR7f / GPCR / receptor / G protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報trace-amine receptor activity / sensory perception of chemical stimulus / Olfactory Signaling Pathway / Activation of the phototransduction cascade / G beta:gamma signalling through PLC beta / Presynaptic function of Kainate receptors / Thromboxane signalling through TP receptor / G-protein activation / G protein-coupled acetylcholine receptor signaling pathway / Activation of G protein gated Potassium channels ...trace-amine receptor activity / sensory perception of chemical stimulus / Olfactory Signaling Pathway / Activation of the phototransduction cascade / G beta:gamma signalling through PLC beta / Presynaptic function of Kainate receptors / Thromboxane signalling through TP receptor / G-protein activation / G protein-coupled acetylcholine receptor signaling pathway / Activation of G protein gated Potassium channels / Inhibition of voltage gated Ca2+ channels via Gbeta/gamma subunits / Prostacyclin signalling through prostacyclin receptor / Glucagon signaling in metabolic regulation / G beta:gamma signalling through CDC42 / ADP signalling through P2Y purinoceptor 12 / G beta:gamma signalling through BTK / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) / Sensory perception of sweet, bitter, and umami (glutamate) taste / photoreceptor disc membrane / Adrenaline,noradrenaline inhibits insulin secretion / Glucagon-type ligand receptors / Vasopressin regulates renal water homeostasis via Aquaporins / G alpha (z) signalling events / cellular response to catecholamine stimulus / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / ADORA2B mediated anti-inflammatory cytokines production / sensory perception of taste / ADP signalling through P2Y purinoceptor 1 / adenylate cyclase-activating dopamine receptor signaling pathway / G beta:gamma signalling through PI3Kgamma / cellular response to prostaglandin E stimulus / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / GPER1 signaling / G-protein beta-subunit binding / Inactivation, recovery and regulation of the phototransduction cascade / heterotrimeric G-protein complex / G alpha (12/13) signalling events / extracellular vesicle / signaling receptor complex adaptor activity / Thrombin signalling through proteinase activated receptors (PARs) / GTPase binding / retina development in camera-type eye / phospholipase C-activating G protein-coupled receptor signaling pathway / Ca2+ pathway / G alpha (i) signalling events / fibroblast proliferation / G alpha (s) signalling events / G alpha (q) signalling events / cell population proliferation / Ras protein signal transduction / Extra-nuclear estrogen signaling / G protein-coupled receptor signaling pathway / lysosomal membrane / GTPase activity / synapse / protein-containing complex binding / signal transduction / extracellular exosome / membrane / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)  | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.92 Å | |||||||||
 データ登録者 データ登録者 | Gusach, A. / Lee, Y. / Edwards, P.C. / Huang, F. / Weyand, S.N. / Tate, C.G. | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Molecular recognition of an aversive odorant by the murine trace amine-associated receptor TAAR7f. 著者: Anastasiia Gusach / Yang Lee / Armin Nikpour Khoshgrudi / Elizaveta Mukhaleva / Ning Ma / Eline J Koers / Qingchao Chen / Patricia C Edwards / Fanglu Huang / Jonathan Kim / Filippo Mancia / ...著者: Anastasiia Gusach / Yang Lee / Armin Nikpour Khoshgrudi / Elizaveta Mukhaleva / Ning Ma / Eline J Koers / Qingchao Chen / Patricia C Edwards / Fanglu Huang / Jonathan Kim / Filippo Mancia / Dmitry B Verprintsev / Nagarajan Vaidehi / Simone N Weyand / Christopher G Tate /   要旨: There are two main families of G protein-coupled receptors that detect odours in humans, the odorant receptors (ORs) and the trace amine-associated receptors (TAARs). Their amino acid sequences are ...There are two main families of G protein-coupled receptors that detect odours in humans, the odorant receptors (ORs) and the trace amine-associated receptors (TAARs). Their amino acid sequences are distinct, with the TAARs being most similar to the aminergic receptors such as those activated by adrenaline, serotonin and histamine. To elucidate the structural determinants of ligand recognition by TAARs, we have determined the cryo-EM structure of a murine receptor, mTAAR7f, coupled to the heterotrimeric G protein G and bound to the odorant N,N-dimethylcyclohexylamine (DMCH) to an overall resolution of 2.9 Å. DMCH is bound in a hydrophobic orthosteric binding site primarily through van der Waals interactions and a strong charge-charge interaction between the tertiary amine of the ligand and an aspartic acid residue. This site is distinct and non-overlapping with the binding site for the odorant propionate in the odorant receptor OR51E2. The structure, in combination with mutagenesis data and molecular dynamics simulations suggests that the activation of the receptor follows a similar pathway to that of the β-adrenoceptors, with the significant difference that DMCH interacts directly with one of the main activation microswitch residues. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8pm2.cif.gz 8pm2.cif.gz | 214.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8pm2.ent.gz pdb8pm2.ent.gz | 160.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8pm2.json.gz 8pm2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8pm2_validation.pdf.gz 8pm2_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8pm2_full_validation.pdf.gz 8pm2_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  8pm2_validation.xml.gz 8pm2_validation.xml.gz | 47.8 KB | 表示 | |
| CIF形式データ |  8pm2_validation.cif.gz 8pm2_validation.cif.gz | 70.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pm/8pm2 https://data.pdbj.org/pub/pdb/validation_reports/pm/8pm2 ftp://data.pdbj.org/pub/pdb/validation_reports/pm/8pm2 ftp://data.pdbj.org/pub/pdb/validation_reports/pm/8pm2 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  17756MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Guanine nucleotide-binding protein ... , 3種, 3分子 ABG
| #1: タンパク質 | 分子量: 28964.734 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: GNAS, GNAS1, GSP / プラスミド: pET15b / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GNAS, GNAS1, GSP / プラスミド: pET15b / 発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 37342.785 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: GNB1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GNB1 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P62873 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P62873 |
| #3: タンパク質 | 分子量: 7845.078 Da / 分子数: 1 / Mutation: C68S / 由来タイプ: 組換発現 / 詳細: There is an engineered mutation C68S introduced / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: GNG2 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GNG2 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P59768 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P59768 |
-抗体 / タンパク質 , 2種, 2分子 NR
| #4: 抗体 | 分子量: 16926.076 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
| #5: タンパク質 | 分子量: 41012.539 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: mTAAR7f sequence contains cleaved protease sites: TEV (S residue on the N terminus) and HRV-3C (C terminus) 由来: (組換発現)   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q5QD08 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q5QD08 |
-非ポリマー , 2種, 2分子 
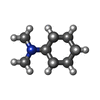

| #6: 化合物 | ChemComp-Y01 / |
|---|---|
| #7: 化合物 | ChemComp-8IA / ~{ |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: A complex of mouse trace-amine associated receptor 7f solubilized in LMNG/CHS bound to N,N-dimethylcyclohexylamine and coupled to: Engineered guanine nucleotide-binding protein G(s) subunit ...名称: A complex of mouse trace-amine associated receptor 7f solubilized in LMNG/CHS bound to N,N-dimethylcyclohexylamine and coupled to: Engineered guanine nucleotide-binding protein G(s) subunit alpha isoform short + Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1 + Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2 + Nanobody 35 タイプ: COMPLEX / Entity ID: #1-#5 / 由来: RECOMBINANT | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) | ||||||||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||||||||||||||
| 試料 | 濃度: 0.8 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||||||||||||||||||
| 試料支持 | 詳細: Forward Power of 38 W, Reflected Power of 2W; Fischione グリッドの材料: GOLD / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: UltrAuFoil R1.2/1.3 | ||||||||||||||||||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 96000 X / 倍率(補正後): 96000 X / 最大 デフォーカス(公称値): 2400 nm / 最小 デフォーカス(公称値): 800 nm / Calibrated defocus min: 2400 nm / 最大 デフォーカス(補正後): 2400 nm / Cs: 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 7.84 sec. / 電子線照射量: 55 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 11157 |
| 画像スキャン | 横: 4096 / 縦: 4096 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | 詳細: Done with cryoSPARC patch motion correction / タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 9199357 / 詳細: Particles were heavily over-picked | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.92 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 172639 / アルゴリズム: FOURIER SPACE / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 詳細: Initial models of heterotrimeric mini-Gs and Nb35 were sourced from PDB 7T9I. A de novo model of TAAR7f was generated from the focused map and protein sequence using ModelAngelo Source name: Other / タイプ: other | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 2.92→2.92 Å / Cor.coef. Fo:Fc: 0.836 / SU B: 13.865 / SU ML: 0.249 / ESU R: 0.346 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 104.792 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 7624 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj























