+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8fyn | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MicroED structure of A2A from plasma milled lamellae | |||||||||
 要素 要素 | Adenosine receptor A2a,Soluble cytochrome b562 | |||||||||
 キーワード キーワード | MEMBRANE PROTEIN / A2A Adenosine Receptor | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound / sensory perception / NGF-independant TRKA activation ...positive regulation of acetylcholine secretion, neurotransmission / positive regulation of circadian sleep/wake cycle, sleep / regulation of norepinephrine secretion / negative regulation of alpha-beta T cell activation / Adenosine P1 receptors / G protein-coupled adenosine receptor activity / G protein-coupled adenosine receptor signaling pathway / response to purine-containing compound / sensory perception / NGF-independant TRKA activation / Surfactant metabolism / positive regulation of urine volume / positive regulation of glutamate secretion / synaptic transmission, dopaminergic / : / inhibitory postsynaptic potential / negative regulation of vascular permeability / type 5 metabotropic glutamate receptor binding / synaptic transmission, cholinergic / blood circulation / response to caffeine / intermediate filament / eating behavior / alpha-actinin binding / presynaptic active zone / regulation of calcium ion transport / membrane depolarization / asymmetric synapse / axolemma / cellular defense response / prepulse inhibition / phagocytosis / presynaptic modulation of chemical synaptic transmission / response to amphetamine / positive regulation of synaptic transmission, glutamatergic / neuron projection morphogenesis / excitatory postsynaptic potential / regulation of mitochondrial membrane potential / positive regulation of long-term synaptic potentiation / central nervous system development / synaptic transmission, glutamatergic / positive regulation of synaptic transmission, GABAergic / locomotory behavior / positive regulation of protein secretion / astrocyte activation / apoptotic signaling pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / electron transport chain / positive regulation of apoptotic signaling pathway / adenylate cyclase-modulating G protein-coupled receptor signaling pathway / negative regulation of inflammatory response / vasodilation / blood coagulation / cell-cell signaling / presynaptic membrane / G alpha (s) signalling events / postsynaptic membrane / negative regulation of neuron apoptotic process / periplasmic space / electron transfer activity / calmodulin binding / inflammatory response / iron ion binding / response to xenobiotic stimulus / negative regulation of cell population proliferation / neuronal cell body / lipid binding / glutamatergic synapse / dendrite / heme binding / protein-containing complex binding / regulation of DNA-templated transcription / apoptotic process / enzyme binding / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / 解像度: 2 Å | |||||||||
 データ登録者 データ登録者 | Martynowycz, M.W. / Shiriaeva, A. / Clabbers, M.T.B. / Nicolas, W.J. / Weaver, S.J. / Hattne, J. / Gonen, T. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: A robust approach for MicroED sample preparation of lipidic cubic phase embedded membrane protein crystals. 著者: Michael W Martynowycz / Anna Shiriaeva / Max T B Clabbers / William J Nicolas / Sara J Weaver / Johan Hattne / Tamir Gonen /  要旨: Crystallizing G protein-coupled receptors (GPCRs) in lipidic cubic phase (LCP) often yields crystals suited for the cryogenic electron microscopy (cryoEM) method microcrystal electron diffraction ...Crystallizing G protein-coupled receptors (GPCRs) in lipidic cubic phase (LCP) often yields crystals suited for the cryogenic electron microscopy (cryoEM) method microcrystal electron diffraction (MicroED). However, sample preparation is challenging. Embedded crystals cannot be targeted topologically. Here, we use an integrated fluorescence light microscope (iFLM) inside of a focused ion beam and scanning electron microscope (FIB-SEM) to identify fluorescently labeled GPCR crystals. Crystals are targeted using the iFLM and LCP is milled using a plasma focused ion beam (pFIB). The optimal ion source for preparing biological lamellae is identified using standard crystals of proteinase K. Lamellae prepared using either argon or xenon produced the highest quality data and structures. MicroED data are collected from the milled lamellae and the structures are determined. This study outlines a robust approach to identify and mill membrane protein crystals for MicroED and demonstrates plasma ion-beam milling is a powerful tool for preparing biological lamellae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8fyn.cif.gz 8fyn.cif.gz | 330.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8fyn.ent.gz pdb8fyn.ent.gz | 229.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8fyn.json.gz 8fyn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8fyn_validation.pdf.gz 8fyn_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8fyn_full_validation.pdf.gz 8fyn_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  8fyn_validation.xml.gz 8fyn_validation.xml.gz | 20.4 KB | 表示 | |
| CIF形式データ |  8fyn_validation.cif.gz 8fyn_validation.cif.gz | 28.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fy/8fyn https://data.pdbj.org/pub/pdb/validation_reports/fy/8fyn ftp://data.pdbj.org/pub/pdb/validation_reports/fy/8fyn ftp://data.pdbj.org/pub/pdb/validation_reports/fy/8fyn | HTTPS FTP |
-関連構造データ
| 関連構造データ |  29586MC  8fyoC  8fypC  8fyqC  8fyrC  8fysC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 49974.281 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: ADORA2A, ADORA2, cybC Homo sapiens (ヒト) / 遺伝子: ADORA2A, ADORA2, cybC発現宿主:  Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾)参照: UniProt: P29274, UniProt: P0ABE7 |
|---|
-非ポリマー , 7種, 200分子 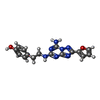











| #2: 化合物 | ChemComp-ZMA / | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
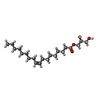
| #3: 化合物 | | #4: 化合物 | ChemComp-OLA / #5: 化合物 | ChemComp-OLC / ( #6: 化合物 | ChemComp-OLB / ( | #7: 化合物 | ChemComp-NA / | #8: 水 | ChemComp-HOH / | |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法: 電子線結晶学 |
|---|---|
| EM実験 | 試料の集合状態: 3D ARRAY / 3次元再構成法: 電子線結晶学 |
- 試料調製
試料調製
| 構成要素 | 名称: A2A BRIL Adenosine receptor / タイプ: COMPLEX / Entity ID: #1 / 由来: NATURAL |
|---|---|
| 分子量 | 値: 0.041 MDa / 実験値: NO |
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 緩衝液 | pH: 4.7 |
| 試料 | 濃度: 25 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES / 詳細: Milled microcrystals |
| 試料支持 | グリッドのタイプ: Quantifoil R2/2 |
| 急速凍結 | 装置: LEICA PLUNGER / 凍結剤: ETHANE / 湿度: 95 % / 凍結前の試料温度: 277 K |
-データ収集
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: DIFFRACTION / 最大 デフォーカス(公称値): 0 nm / 最小 デフォーカス(公称値): 0 nm / Cs: 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: BASIC |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER 最高温度: 90 K / 最低温度: 77 K |
| 撮影 | 平均露光時間: 0.5 sec. / 電子線照射量: 0.001 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) Num. of diffraction images: 840 / 撮影したグリッド数: 1 / 実像数: 1 |
| 画像スキャン | サンプリングサイズ: 28 µm / 横: 2048 / 縦: 2048 |
| EM回折 | カメラ長: 1202 mm / Tilt angle list: -30,30 |
| EM回折 シェル | 解像度: 0.87→0.9 Å / フーリエ空間範囲: 37.64 % / 多重度: 2.1 / 構造因子数: 2783 / 位相残差: 30 ° |
| EM回折 統計 | フーリエ空間範囲: 87.58 % / 再高解像度: 0.87 Å / 測定した強度の数: 569407 / 構造因子数: 64974 / 位相誤差: 30 ° / 位相誤差の除外基準: None / Rmerge: 0.236 / Rsym: 0.073 |
| 反射 | Biso Wilson estimate: 12.54 Å2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア | 名称: AIMLESS / カテゴリ: crystallography merging | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 画像処理 | 詳細: Binned by 2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| EM 3D crystal entity | ∠α: 90 ° / ∠β: 90 ° / ∠γ: 90 ° / A: 39.08 Å / B: 177.51 Å / C: 139.29 Å / 空間群名: 20 / 空間群番号: 20 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES / アルゴリズム: FOURIER SPACE / 対称性のタイプ: 3D CRYSTAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 12.54 / プロトコル: AB INITIO MODEL / 空間: RECIPROCAL / Target criteria: Maximum likelihood | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 2→41.14 Å / SU ML: 0.2918 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 28.6157 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.6 Å / VDWプローブ半径: 0.9 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 23.1 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→41.14 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj






















