+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8fne | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
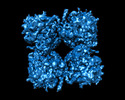
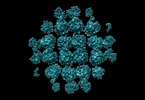
| タイトル | phiPA3 PhuN Tetramer, p2 | |||||||||
 要素 要素 | Maltose/maltodextrin-binding periplasmic protein, PhuN | |||||||||
 キーワード キーワード | VIRAL PROTEIN / phiPA3 protein / shell protein / PhuN / phage nucleus | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報detection of maltose stimulus / maltose transport complex / carbohydrate transport / carbohydrate transmembrane transporter activity / maltose binding / maltose transport / maltodextrin transmembrane transport / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / ATP-binding cassette (ABC) transporter complex / cell chemotaxis ...detection of maltose stimulus / maltose transport complex / carbohydrate transport / carbohydrate transmembrane transporter activity / maltose binding / maltose transport / maltodextrin transmembrane transport / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / ATP-binding cassette (ABC) transporter complex / cell chemotaxis / outer membrane-bounded periplasmic space / host cell cytoplasm / periplasmic space / DNA damage response / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Pseudomonas phage PhiPA3 (ファージ) Pseudomonas phage PhiPA3 (ファージ) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Nieweglowska, E.S. / Brilot, A.F. / Mendez-Moran, M. / Kokontis, C. / Baek, M. / Li, J. / Cheng, Y. / Baker, D. / Bondy-Denomy, J. / Agard, D.A. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: The ϕPA3 phage nucleus is enclosed by a self-assembling 2D crystalline lattice. 著者: Eliza S Nieweglowska / Axel F Brilot / Melissa Méndez-Moran / Claire Kokontis / Minkyung Baek / Junrui Li / Yifan Cheng / David Baker / Joseph Bondy-Denomy / David A Agard /  要旨: To protect themselves from host attack, numerous jumbo bacteriophages establish a phage nucleus-a micron-scale, proteinaceous structure encompassing the replicating phage DNA. Bacteriophage and host ...To protect themselves from host attack, numerous jumbo bacteriophages establish a phage nucleus-a micron-scale, proteinaceous structure encompassing the replicating phage DNA. Bacteriophage and host proteins associated with replication and transcription are concentrated inside the phage nucleus while other phage and host proteins are excluded, including CRISPR-Cas and restriction endonuclease host defense systems. Here, we show that nucleus fragments isolated from ϕPA3 infected Pseudomonas aeruginosa form a 2-dimensional lattice, having p2 or p4 symmetry. We further demonstrate that recombinantly purified primary Phage Nuclear Enclosure (PhuN) protein spontaneously assembles into similar 2D sheets with p2 and p4 symmetry. We resolve the dominant p2 symmetric state to 3.9 Å by cryo-EM. Our structure reveals a two-domain core, organized into quasi-symmetric tetramers. Flexible loops and termini mediate adaptable inter-tetramer contacts that drive subunit assembly into a lattice and enable the adoption of different symmetric states. While the interfaces between subunits are mostly well packed, two are open, forming channels that likely have functional implications for the transport of proteins, mRNA, and small molecules. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8fne.cif.gz 8fne.cif.gz | 799.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8fne.ent.gz pdb8fne.ent.gz | 607 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8fne.json.gz 8fne.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8fne_validation.pdf.gz 8fne_validation.pdf.gz | 1019.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8fne_full_validation.pdf.gz 8fne_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  8fne_validation.xml.gz 8fne_validation.xml.gz | 64.3 KB | 表示 | |
| CIF形式データ |  8fne_validation.cif.gz 8fne_validation.cif.gz | 99.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fn/8fne https://data.pdbj.org/pub/pdb/validation_reports/fn/8fne ftp://data.pdbj.org/pub/pdb/validation_reports/fn/8fne ftp://data.pdbj.org/pub/pdb/validation_reports/fn/8fne | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 110126.078 Da / 分子数: 8 / 由来タイプ: 組換発現 由来: (組換発現)   Pseudomonas phage PhiPA3 (ファージ) Pseudomonas phage PhiPA3 (ファージ)遺伝子: malE, b4034, JW3994, 053 / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: 2D ARRAY / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Core tetramer assembly (p2) of the phiPA3 bacteriophage PhuN protein タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | ||||||||||||||||||||||||||||
| 由来(天然) | 生物種:  Pseudomonas phage PhiPA3 (ファージ) Pseudomonas phage PhiPA3 (ファージ) | ||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:  | ||||||||||||||||||||||||||||
| 緩衝液 | pH: 6.5 詳細: 0.25 cOmplete Protease Inhibitor Tablet also included | ||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 283.15 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 105000 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 800 nm / Cs: 2.7 mm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 67 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C2 (2回回転対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 29303 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT 詳細: The initial model was generated using AlphaFold based on sequence alone. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | Source name: AlphaFold / タイプ: in silico model |
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj


