+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7x5k | ||||||
|---|---|---|---|---|---|---|---|
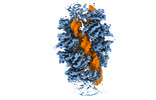
| タイトル | Tir-dsDNA complex, the initial binding state | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE/DNA / plant innate immune receptor / nucleic acids / 2' / 3'cNMP / HYDROLASE-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報induction of programmed cell death / NADP catabolic process / cGMP biosynthetic process / NAD catabolic process / cAMP biosynthetic process / DNA catabolic process / RNA catabolic process / cAMP binding / ADP binding / defense response ...induction of programmed cell death / NADP catabolic process / cGMP biosynthetic process / NAD catabolic process / cAMP biosynthetic process / DNA catabolic process / RNA catabolic process / cAMP binding / ADP binding / defense response / double-stranded RNA binding / double-stranded DNA binding / hydrolase activity / signal transduction 類似検索 - 分子機能 | ||||||
| 生物種 | DNA molecule (その他) Linum usitatissimum (アマ) Linum usitatissimum (アマ) | ||||||
| 手法 | 電子顕微鏡法 / らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | ||||||
 データ登録者 データ登録者 | Tan, Y. / Xu, C. / Yu, D. / Song, W. / Wu, B. / Schulze-Lefert, P. / Chai, J. | ||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| ||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: TIR domains of plant immune receptors are 2',3'-cAMP/cGMP synthetases mediating cell death. 著者: Dongli Yu / Wen Song / Eddie Yong Jun Tan / Li Liu / Yu Cao / Jan Jirschitzka / Ertong Li / Elke Logemann / Chenrui Xu / Shijia Huang / Aolin Jia / Xiaoyu Chang / Zhifu Han / Bin Wu / Paul ...著者: Dongli Yu / Wen Song / Eddie Yong Jun Tan / Li Liu / Yu Cao / Jan Jirschitzka / Ertong Li / Elke Logemann / Chenrui Xu / Shijia Huang / Aolin Jia / Xiaoyu Chang / Zhifu Han / Bin Wu / Paul Schulze-Lefert / Jijie Chai /    要旨: 2',3'-cAMP is a positional isomer of the well-established second messenger 3',5'-cAMP, but little is known about the biology of this noncanonical cyclic nucleotide monophosphate (cNMP). ...2',3'-cAMP is a positional isomer of the well-established second messenger 3',5'-cAMP, but little is known about the biology of this noncanonical cyclic nucleotide monophosphate (cNMP). Toll/interleukin-1 receptor (TIR) domains of nucleotide-binding leucine-rich repeat (NLR) immune receptors have the NADase function necessary but insufficient to activate plant immune responses. Here, we show that plant TIR proteins, besides being NADases, act as 2',3'-cAMP/cGMP synthetases by hydrolyzing RNA/DNA. Structural data show that a TIR domain adopts distinct oligomers with mutually exclusive NADase and synthetase activity. Mutations specifically disrupting the synthetase activity abrogate TIR-mediated cell death in Nicotiana benthamiana (Nb), supporting an important role for these cNMPs in TIR signaling. Furthermore, the Arabidopsis negative regulator of TIR-NLR signaling, NUDT7, displays 2',3'-cAMP/cGMP but not 3',5'-cAMP/cGMP phosphodiesterase activity and suppresses cell death activity of TIRs in Nb. Our study identifies a family of 2',3'-cAMP/cGMP synthetases and establishes a critical role for them in plant immune responses. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7x5k.cif.gz 7x5k.cif.gz | 564.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7x5k.ent.gz pdb7x5k.ent.gz | 465.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7x5k.json.gz 7x5k.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7x5k_validation.pdf.gz 7x5k_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7x5k_full_validation.pdf.gz 7x5k_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  7x5k_validation.xml.gz 7x5k_validation.xml.gz | 93.2 KB | 表示 | |
| CIF形式データ |  7x5k_validation.cif.gz 7x5k_validation.cif.gz | 122 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/x5/7x5k https://data.pdbj.org/pub/pdb/validation_reports/x5/7x5k ftp://data.pdbj.org/pub/pdb/validation_reports/x5/7x5k ftp://data.pdbj.org/pub/pdb/validation_reports/x5/7x5k | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 13215.611 Da / 分子数: 2 / 由来タイプ: 組換発現 詳細: Residues DA B 12 and DT B 13 that are next to each other in the sample sequence are not properly linked: distance between O3' and P is 4.53. This out of range distance is due to an ...詳細: Residues DA B 12 and DT B 13 that are next to each other in the sample sequence are not properly linked: distance between O3' and P is 4.53. This out of range distance is due to an intermediate state of a dsDNA hydrolysis process. Likewise, for the same reason, out of range distances are shown at the same position of chain Q. 由来: (組換発現) DNA molecule (その他) / 発現宿主: synthetic construct (人工物) #2: DNA鎖 | 分子量: 13242.652 Da / 分子数: 2 / 由来タイプ: 組換発現 詳細: Residues DA E 12 and DT E 13 that are next to each other in the sample sequence are not properly linked: distance between O3' and P is 3.89. This out of range distance is due to an ...詳細: Residues DA E 12 and DT E 13 that are next to each other in the sample sequence are not properly linked: distance between O3' and P is 3.89. This out of range distance is due to an intermediate state of a dsDNA hydrolysis process. Likewise, for the same reason, out of range distances are shown at the same position of chain S. 由来: (組換発現) DNA molecule (その他) / 発現宿主: synthetic construct (人工物) #3: タンパク質 | 分子量: 23532.344 Da / 分子数: 16 / Mutation: E197G / 由来タイプ: 組換発現 / 詳細: L7 Tir domain / 由来: (組換発現)  Linum usitatissimum (アマ) / 発現宿主: Linum usitatissimum (アマ) / 発現宿主:  構成要素の詳細 | Residues DA B 12 and DT B 13 that are next to each other in the sample sequence are not properly ...Residues DA B 12 and DT B 13 that are next to each other in the sample sequence are not properly linked: distance between O3' and P is 4.53. This out of range distance is due to an intermediate state of a dsDNA hydrolysis process. Likewise, for the same reason, out of range distances are shown at the same position of chain E, Q and S. | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: らせん対称体再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 20 kDa/nm / 実験値: NO | ||||||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||||||
| 緩衝液 | pH: 7 | ||||||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||||||
| 急速凍結 | 凍結剤: METHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1000 nm / アライメント法: BASIC |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.18rc5_3822: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア | 名称: RELION / バージョン: 3.08 / カテゴリ: 3次元再構成 | ||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| らせん対称 | 回転角度/サブユニット: 159.2 ° / 軸方向距離/サブユニット: 16.56 Å / らせん対称軸の対称性: C1 | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 228796 / 対称性のタイプ: HELICAL | ||||||||||||||||||||||||
| 原子モデル構築 | B value: 215 / プロトコル: RIGID BODY FIT / 空間: REAL | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj








































