+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6ovh | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Bimetallic dodecameric cage design 3 (BMC3) from cytochrome cb562 | |||||||||||||||
 要素 要素 | Soluble cytochrome b562 | |||||||||||||||
 キーワード キーワード | METAL BINDING PROTEIN / Supramolecular assembly / protein cage / bimetallic / metal binding / hydroxamic acid | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報electron transport chain / periplasmic space / electron transfer activity / iron ion binding / heme binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||||||||
| Model details | Keywords: Supramolecular assembly, protein cage, bimetallic, metal binding | |||||||||||||||
 データ登録者 データ登録者 | Golub, E. / Subramanian, R.H. / Yan, X. / Alberstein, R.G. / Tezcan, F.A. | |||||||||||||||
| 資金援助 |  米国, European Union, 米国, European Union,  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Constructing protein polyhedra via orthogonal chemical interactions. 著者: Eyal Golub / Rohit H Subramanian / Julian Esselborn / Robert G Alberstein / Jake B Bailey / Jerika A Chiong / Xiaodong Yan / Timothy Booth / Timothy S Baker / F Akif Tezcan /  要旨: Many proteins exist naturally as symmetrical homooligomers or homopolymers. The emergent structural and functional properties of such protein assemblies have inspired extensive efforts in ...Many proteins exist naturally as symmetrical homooligomers or homopolymers. The emergent structural and functional properties of such protein assemblies have inspired extensive efforts in biomolecular design. As synthesized by ribosomes, proteins are inherently asymmetric. Thus, they must acquire multiple surface patches that selectively associate to generate the different symmetry elements needed to form higher-order architectures-a daunting task for protein design. Here we address this problem using an inorganic chemical approach, whereby multiple modes of protein-protein interactions and symmetry are simultaneously achieved by selective, 'one-pot' coordination of soft and hard metal ions. We show that a monomeric protein (protomer) appropriately modified with biologically inspired hydroxamate groups and zinc-binding motifs assembles through concurrent Fe and Zn coordination into discrete dodecameric and hexameric cages. Our cages closely resemble natural polyhedral protein architectures and are, to our knowledge, unique among designed systems in that they possess tightly packed shells devoid of large apertures. At the same time, they can assemble and disassemble in response to diverse stimuli, owing to their heterobimetallic construction on minimal interprotein-bonding footprints. With stoichiometries ranging from [2 Fe:9 Zn:6 protomers] to [8 Fe:21 Zn:12 protomers], these protein cages represent some of the compositionally most complex protein assemblies-or inorganic coordination complexes-obtained by design. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6ovh.cif.gz 6ovh.cif.gz | 256.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6ovh.ent.gz pdb6ovh.ent.gz | 217.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6ovh.json.gz 6ovh.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6ovh_validation.pdf.gz 6ovh_validation.pdf.gz | 1.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6ovh_full_validation.pdf.gz 6ovh_full_validation.pdf.gz | 1.8 MB | 表示 | |
| XML形式データ |  6ovh_validation.xml.gz 6ovh_validation.xml.gz | 41.8 KB | 表示 | |
| CIF形式データ |  6ovh_validation.cif.gz 6ovh_validation.cif.gz | 66.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ov/6ovh https://data.pdbj.org/pub/pdb/validation_reports/ov/6ovh ftp://data.pdbj.org/pub/pdb/validation_reports/ov/6ovh ftp://data.pdbj.org/pub/pdb/validation_reports/ov/6ovh | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 1種, 12分子 ABCDEFGHIJKL
| #1: タンパク質 | 分子量: 11809.307 Da / 分子数: 12 変異: D5H,E8H,V16H,A24T,Q25T,R34Q,L38Q,Q41W,K42S,K59S,H63C,D66W,I67E,V69I,D73N,D74A,K77H,N80K,E81Q,G82C,R98C,Y101C 由来タイプ: 組換発現 詳細: pET20b for expression of BMC3 described here with background of pEC86 to provide machinery for c-type linkage of heme. 由来: (組換発現)  詳細 (発現宿主): pET20b for expression of BMC3 described here with background of pEC86 to provide machinery for c-type linkage of heme. 発現宿主:  |
|---|
-非ポリマー , 5種, 239分子 
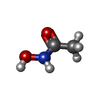







| #2: 化合物 | ChemComp-HEC / #3: 化合物 | ChemComp-HAE / #4: 化合物 | ChemComp-ZN / #5: 化合物 | ChemComp-FE / #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Bimetallic dodecameric cage 3 (BMC3) / タイプ: COMPLEX 詳細: Cryo-EM reconstruction of self-assembled BMC3 dodecameric cages Entity ID: #1 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.15 MDa / 実験値: YES |
| 由来(天然) | 生物種:  |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 8.5 |
| 緩衝液成分 | 濃度: 20 mM / 名称: Tris |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES 詳細: Protein solutions containing 20 micromolar BMC3 in 20 mM Tris (pH 8.5) were incubated with [FeSO4] = 20 micromolar, [ZnCl2] = 60 micromolar for 2-3 h to form cages. Samples were concentrated ...詳細: Protein solutions containing 20 micromolar BMC3 in 20 mM Tris (pH 8.5) were incubated with [FeSO4] = 20 micromolar, [ZnCl2] = 60 micromolar for 2-3 h to form cages. Samples were concentrated 10 fold prior to grid preparation. |
| 試料支持 | 詳細: The grid was glow discharged at 20 mA for 30 s. / グリッドの材料: COPPER / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 平均露光時間: 10 sec. / 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 4672 |
| 画像スキャン | 動画フレーム数/画像: 50 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 805156 | ||||||||||||||||||||||||
| 対称性 | 点対称性: T (正4面体型対称) | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 25391 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL 詳細: Symmetry mates were generated from the atomic model to build the dodecameric cage. Chain IDs were re-assigned to ascend from A-L. The cage PDB was manually fit to the EM density map in UCSF ...詳細: Symmetry mates were generated from the atomic model to build the dodecameric cage. Chain IDs were re-assigned to ascend from A-L. The cage PDB was manually fit to the EM density map in UCSF Chimera and refined using phenix.real_space_refine | ||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 6OT7 | ||||||||||||||||||||||||
| 精密化 | 立体化学のターゲット値: CDL v1.2 | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

















