+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4bkk | ||||||
|---|---|---|---|---|---|---|---|
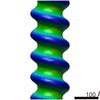
| タイトル | The Respiratory Syncytial Virus nucleoprotein-RNA complex forms a left-handed helical nucleocapsid. | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | VIRAL PROTEIN/RNA / VIRAL PROTEIN-RNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Respiratory syncytial virus genome transcription / Translation of respiratory syncytial virus mRNAs / symbiont-mediated suppression of host PKR/eIFalpha signaling / protein serine/threonine kinase inhibitor activity / helical viral capsid / Respiratory syncytial virus genome replication / RSV-host interactions / Maturation of hRSV A proteins / Assembly and release of respiratory syncytial virus (RSV) virions / Respiratory syncytial virus (RSV) attachment and entry ...Respiratory syncytial virus genome transcription / Translation of respiratory syncytial virus mRNAs / symbiont-mediated suppression of host PKR/eIFalpha signaling / protein serine/threonine kinase inhibitor activity / helical viral capsid / Respiratory syncytial virus genome replication / RSV-host interactions / Maturation of hRSV A proteins / Assembly and release of respiratory syncytial virus (RSV) virions / Respiratory syncytial virus (RSV) attachment and entry / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / PKR-mediated signaling / Evasion by RSV of host interferon responses / viral capsid / viral nucleocapsid / symbiont-mediated suppression of host NF-kappaB cascade / host cell cytoplasm / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / ribonucleoprotein complex / RNA binding 類似検索 - 分子機能 | ||||||
| 生物種 |  HUMAN RESPIRATORY SYNCYTIAL VIRUS A STRAIN LONG (ウイルス) HUMAN RESPIRATORY SYNCYTIAL VIRUS A STRAIN LONG (ウイルス) | ||||||
| 手法 | 電子顕微鏡法 / 電子線トモグラフィー法 / ネガティブ染色法 | ||||||
 データ登録者 データ登録者 | Bakker, S.E. / Duquerroy, S. / Galloux, M. / Loney, C. / Conner, E. / Eleouet, J.F. / Rey, F.A. / Bhella, D. | ||||||
 引用 引用 |  ジャーナル: J Gen Virol / 年: 2013 ジャーナル: J Gen Virol / 年: 2013タイトル: The respiratory syncytial virus nucleoprotein-RNA complex forms a left-handed helical nucleocapsid. 著者: Saskia E Bakker / Stéphane Duquerroy / Marie Galloux / Colin Loney / Edward Conner / Jean-François Eléouët / Félix A Rey / David Bhella /   要旨: Respiratory syncytial virus (RSV) is an important human pathogen. Its nucleocapsid (NC), which comprises the negative sense RNA viral genome coated by the viral nucleoprotein N, is a critical ...Respiratory syncytial virus (RSV) is an important human pathogen. Its nucleocapsid (NC), which comprises the negative sense RNA viral genome coated by the viral nucleoprotein N, is a critical assembly that serves as template for both mRNA synthesis and genome replication. We have previously described the X-ray structure of an NC-like structure: a decameric ring formed of N-RNA that mimics one turn of the helical NC. In the absence of experimental data we had hypothesized that the NC helix would be right-handed, as the N-N contacts in the ring appeared to more easily adapt to that conformation. We now unambiguously show that the RSV NC is a left-handed helix. We further show that the contacts in the ring can be distorted to maintain key N-N-protein interactions in a left-handed helix, and discuss the implications of the resulting atomic model of the helical NC for viral replication and transcription. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4bkk.cif.gz 4bkk.cif.gz | 1.6 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4bkk.ent.gz pdb4bkk.ent.gz | 1.3 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4bkk.json.gz 4bkk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4bkk_validation.pdf.gz 4bkk_validation.pdf.gz | 451.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4bkk_full_validation.pdf.gz 4bkk_full_validation.pdf.gz | 512 KB | 表示 | |
| XML形式データ |  4bkk_validation.xml.gz 4bkk_validation.xml.gz | 167.8 KB | 表示 | |
| CIF形式データ |  4bkk_validation.cif.gz 4bkk_validation.cif.gz | 281.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bk/4bkk https://data.pdbj.org/pub/pdb/validation_reports/bk/4bkk ftp://data.pdbj.org/pub/pdb/validation_reports/bk/4bkk ftp://data.pdbj.org/pub/pdb/validation_reports/bk/4bkk | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 49089.227 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  HUMAN RESPIRATORY SYNCYTIAL VIRUS A STRAIN LONG (ウイルス) HUMAN RESPIRATORY SYNCYTIAL VIRUS A STRAIN LONG (ウイルス)細胞株 (発現宿主): Sf21 発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 43507.848 Da / 分子数: 23 / 由来タイプ: 組換発現 由来: (組換発現)  HUMAN RESPIRATORY SYNCYTIAL VIRUS A STRAIN LONG (ウイルス) HUMAN RESPIRATORY SYNCYTIAL VIRUS A STRAIN LONG (ウイルス)細胞株 (発現宿主): Sf21 発現宿主:  参照: UniProt: P03418 |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: 電子線トモグラフィー法 |
- 試料調製
試料調製
| 構成要素 | 名称: PURIFIED RECOMBINANT RSV NUCLEOCAPSIDS. / タイプ: VIRUS |
|---|---|
| 緩衝液 | 名称: 50 MM TRIS-HCL, PH 7.4, 150 MM NACL, 10 MM EDTA / pH: 7.4 / 詳細: 50 MM TRIS-HCL, PH 7.4, 150 MM NACL, 10 MM EDTA |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: YES / 凍結: NO |
| 染色 | タイプ: NEGATIVE / 染色剤: Ammonium Molybdate |
| 試料支持 | 詳細: HOLEY CARBON |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: JEOL 2200FSC / 日付: 2010年7月15日 詳細: ROOM TEMPERATURE STAINED GRIDS IMAGED AT LN2 TEMPERATURES. |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 40000 X / 最大 デフォーカス(公称値): 4000 nm / 最小 デフォーカス(公称値): 2000 nm / Cs: 2 mm |
| 試料ホルダ | 傾斜角・最大: 60 ° / 傾斜角・最小: -60 ° |
| 撮影 | 電子線照射量: 131 e/Å2 フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) |
| 画像スキャン | デジタル画像の数: 9 |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 3次元再構成 | 手法: SUBTOMOGRAM AVERAGING / 粒子像の数: 911 / ピクセルサイズ(公称値): 6.5 Å 詳細: SUBTOMOGRAM AVERAGING. NOTE THE COORDINATES ARE BASED ON MODELLING. SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-2369. (DEPOSITION ID: 11640). 対称性のタイプ: HELICAL | ||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 詳細: METHOD--MODELLING REFINEMENT PROTOCOL--MODEL | ||||||||||||
| 原子モデル構築 | PDB-ID: 2WJ8 Accession code: 2WJ8 / Source name: PDB / タイプ: experimental model | ||||||||||||
| 精密化ステップ | サイクル: LAST
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj






























