+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9333 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacillus PS3 ATP synthase class 1 | |||||||||
 マップデータ マップデータ | ATP synthase class 1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacterial ATP synthase / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proton motive force-driven plasma membrane ATP synthesis / proton-transporting ATP synthase complex, coupling factor F(o) / proton-transporting ATP synthase complex, catalytic core F(1) / H+-transporting two-sector ATPase / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / ADP binding / lipid binding / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
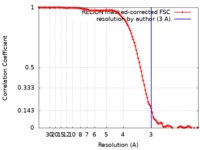
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Guo H / Rubinstein JL | |||||||||
| 資金援助 |  カナダ, カナダ,  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Structure of a bacterial ATP synthase. 著者: Hui Guo / Toshiharu Suzuki / John L Rubinstein /   要旨: ATP synthases produce ATP from ADP and inorganic phosphate with energy from a transmembrane proton motive force. Bacterial ATP synthases have been studied extensively because they are the simplest ...ATP synthases produce ATP from ADP and inorganic phosphate with energy from a transmembrane proton motive force. Bacterial ATP synthases have been studied extensively because they are the simplest form of the enzyme and because of the relative ease of genetic manipulation of these complexes. We expressed the PS3 ATP synthase in , purified it, and imaged it by cryo-EM, allowing us to build atomic models of the complex in three rotational states. The position of subunit shows how it is able to inhibit ATP hydrolysis while allowing ATP synthesis. The architecture of the membrane region shows how the simple bacterial ATP synthase is able to perform the same core functions as the equivalent, but more complicated, mitochondrial complex. The structures reveal the path of transmembrane proton translocation and provide a model for understanding decades of biochemical analysis interrogating the roles of specific residues in the enzyme. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9333.map.gz emd_9333.map.gz | 6.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9333-v30.xml emd-9333-v30.xml emd-9333.xml emd-9333.xml | 29.4 KB 29.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9333_fsc.xml emd_9333_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9333.png emd_9333.png | 60.8 KB | ||
| マスクデータ |  emd_9333_msk_1.map emd_9333_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-9333.cif.gz emd-9333.cif.gz | 7.8 KB | ||
| その他 |  emd_9333_half_map_1.map.gz emd_9333_half_map_1.map.gz emd_9333_half_map_2.map.gz emd_9333_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9333 http://ftp.pdbj.org/pub/emdb/structures/EMD-9333 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9333 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9333 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9333_validation.pdf.gz emd_9333_validation.pdf.gz | 829.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9333_full_validation.pdf.gz emd_9333_full_validation.pdf.gz | 828.7 KB | 表示 | |
| XML形式データ |  emd_9333_validation.xml.gz emd_9333_validation.xml.gz | 18.9 KB | 表示 | |
| CIF形式データ |  emd_9333_validation.cif.gz emd_9333_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9333 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9333 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9333 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9333 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6n2yMC  9327C  9334C  9335C  9336C  9337C  9338C  6n2dC  6n2zC  6n30C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9333.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9333.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ATP synthase class 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
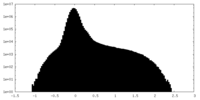
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9333_msk_1.map emd_9333_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
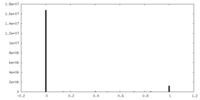
| 密度ヒストグラム |
-ハーフマップ: ATP synthase class 1
| ファイル | emd_9333_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ATP synthase class 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
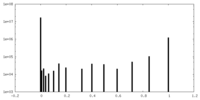
| 密度ヒストグラム |
-ハーフマップ: ATP synthase class 1
| ファイル | emd_9333_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ATP synthase class 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
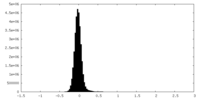
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Bacillus PS3 ATP synthase class 1
+超分子 #1: Bacillus PS3 ATP synthase class 1
+分子 #1: ATP synthase subunit alpha
+分子 #2: ATP synthase subunit beta
+分子 #3: ATP synthase gamma chain
+分子 #4: ATP synthase epsilon chain
+分子 #5: ATP synthase subunit delta
+分子 #6: ATP synthase subunit b
+分子 #7: ATP synthase subunit b
+分子 #8: ATP synthase subunit a
+分子 #9: ATP synthase subunit c
+分子 #10: ADENOSINE-5'-TRIPHOSPHATE
+分子 #11: MAGNESIUM ION
+分子 #12: PHOSPHATE ION
+分子 #13: ADENOSINE-5'-DIPHOSPHATE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均露光時間: 60.0 sec. / 平均電子線量: 0.71 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 132075 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6n2y: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)