[English] 日本語
 Yorodumi
Yorodumi- EMDB-8728: CryoEM Structure of the Zinc Transporter YiiP from helical crystals -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-8728 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | CryoEM Structure of the Zinc Transporter YiiP from helical crystals | |||||||||
 Map data Map data | primary map | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | zinc antiporter / membrane protein / cation diffusion facilitator / metal transport / helical crystals / Structural Genomics / PSI-Biology / Transcontinental EM Initiative for Membrane Protein Structure / TEMIMPS | |||||||||
| Function / homology |  Function and homology information Function and homology informationzinc efflux antiporter activity / cadmium ion transmembrane transporter activity / ferrous iron transmembrane transporter activity / intracellular zinc ion homeostasis / metal ion binding / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Shewanella oneidensis MR-1 (bacteria) Shewanella oneidensis MR-1 (bacteria) | |||||||||
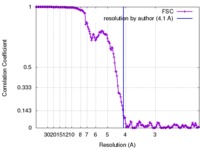
| Method | helical reconstruction / cryo EM / Resolution: 4.1 Å | |||||||||
 Authors Authors | Coudray N | |||||||||
| Funding support |  United States, 2 items United States, 2 items
| |||||||||
 Citation Citation |  Journal: Proc Natl Acad Sci U S A / Year: 2018 Journal: Proc Natl Acad Sci U S A / Year: 2018Title: Structural basis for the alternating access mechanism of the cation diffusion facilitator YiiP. Authors: Maria Luisa Lopez-Redondo / Nicolas Coudray / Zhening Zhang / John Alexopoulos / David L Stokes /  Abstract: YiiP is a dimeric antiporter from the cation diffusion facilitator family that uses the proton motive force to transport Zn across bacterial membranes. Previous work defined the atomic structure of ...YiiP is a dimeric antiporter from the cation diffusion facilitator family that uses the proton motive force to transport Zn across bacterial membranes. Previous work defined the atomic structure of an outward-facing conformation, the location of several Zn binding sites, and hydrophobic residues that appear to control access to the transport sites from the cytoplasm. A low-resolution cryo-EM structure revealed changes within the membrane domain that were associated with the alternating access mechanism for transport. In the current work, the resolution of this cryo-EM structure has been extended to 4.1 Å. Comparison with the X-ray structure defines the differences between inward-facing and outward-facing conformations at an atomic level. These differences include rocking and twisting of a four-helix bundle that harbors the Zn transport site and controls its accessibility within each monomer. As previously noted, membrane domains are closely associated in the dimeric structure from cryo-EM but dramatically splayed apart in the X-ray structure. Cysteine crosslinking was used to constrain these membrane domains and to show that this large-scale splaying was not necessary for transport activity. Furthermore, dimer stability was not compromised by mutagenesis of elements in the cytoplasmic domain, suggesting that the extensive interface between membrane domains is a strong determinant of dimerization. As with other secondary transporters, this interface could provide a stable scaffold for movements of the four-helix bundle that confers alternating access of these ions to opposite sides of the membrane. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_8728.map.gz emd_8728.map.gz | 980.5 KB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-8728-v30.xml emd-8728-v30.xml emd-8728.xml emd-8728.xml | 16.6 KB 16.6 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_8728_fsc.xml emd_8728_fsc.xml | 15.5 KB | Display |  FSC data file FSC data file |
| Images |  emd_8728.png emd_8728.png | 173.2 KB | ||
| Filedesc metadata |  emd-8728.cif.gz emd-8728.cif.gz | 6.6 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8728 http://ftp.pdbj.org/pub/emdb/structures/EMD-8728 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8728 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8728 | HTTPS FTP |
-Related structure data
| Related structure data |  5vrfMC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_8728.map.gz / Format: CCP4 / Size: 8.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_8728.map.gz / Format: CCP4 / Size: 8.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
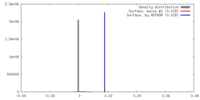
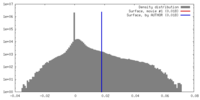
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : YiiP dimer in an inward-facing conformation
| Entire | Name: YiiP dimer in an inward-facing conformation |
|---|---|
| Components |
|
-Supramolecule #1: YiiP dimer in an inward-facing conformation
| Supramolecule | Name: YiiP dimer in an inward-facing conformation / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  Shewanella oneidensis MR-1 (bacteria) Shewanella oneidensis MR-1 (bacteria) |
| Molecular weight | Theoretical: 60 KDa |
-Macromolecule #1: Cadmium and zinc efflux pump FieF
| Macromolecule | Name: Cadmium and zinc efflux pump FieF / type: protein_or_peptide / ID: 1 / Number of copies: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Shewanella oneidensis MR-1 (bacteria) / Strain: MR-1 Shewanella oneidensis MR-1 (bacteria) / Strain: MR-1 |
| Molecular weight | Theoretical: 32.485211 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MTQTSQYDFW VKLASRASVA TALTLITIKL LAWLYSGSAS MLASLTDSFA DTLASIINFI AIRYAIVPAD HDHRYGHGKA EPLAALAQS AFIMGSAFLL LFYGGERLLN PSPVENATLG VVVSVVAIVL TLALVLLQKR ALAATNSTVV EADSLHYKSD L FLNAAVLL ...String: MTQTSQYDFW VKLASRASVA TALTLITIKL LAWLYSGSAS MLASLTDSFA DTLASIINFI AIRYAIVPAD HDHRYGHGKA EPLAALAQS AFIMGSAFLL LFYGGERLLN PSPVENATLG VVVSVVAIVL TLALVLLQKR ALAATNSTVV EADSLHYKSD L FLNAAVLL ALVLSQYGWW WADGLFAVLI ACYIGQQAFD LGYRSIQALL DRELDEDTRQ RIKLIAKEDP RVLGLHDLRT RQ AGKTVFI QFHLELDGNL SLNEAHSITD TTGLRVKAAF EDAEVIIHQD PVQVEPTTQ UniProtKB: Cation-efflux pump FieF |
-Macromolecule #2: ZINC ION
| Macromolecule | Name: ZINC ION / type: ligand / ID: 2 / Number of copies: 8 / Formula: ZN |
|---|---|
| Molecular weight | Theoretical: 65.409 Da |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | helical reconstruction |
| Aggregation state | helical array |
- Sample preparation
Sample preparation
| Concentration | 0.6 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7 Component:
Details: Buffer was changed twice per day. | ||||||||||||
| Grid | Model: EMS / Material: COPPER / Mesh: 400 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 30 sec. / Pretreatment - Atmosphere: OTHER | ||||||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 90 % / Instrument: HOMEMADE PLUNGER | ||||||||||||
| Details | The protein was purified in 0.2% n-dodecyl beta-D-maltoside |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Detector mode: COUNTING / Digitization - Dimensions - Width: 3710 pixel / Digitization - Dimensions - Height: 3838 pixel / Digitization - Frames/image: 2-16 / Number grids imaged: 3 / Number real images: 2743 / Average exposure time: 10.0 sec. / Average electron dose: 70.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source: LAB6 |
| Electron optics | Calibrated defocus max: 5.4 µm / Calibrated defocus min: 1.0 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.2 µm / Nominal magnification: 22500 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Initial model |
| ||||||
|---|---|---|---|---|---|---|---|
| Details | The residues were manually adjusted in COOT relying on the large side-chain residues, and the whole system was further optimized using the real_space_refine algorithm in Phenix to ensure proper fit. The conformation was subjected to 13 rounds of COOT/Phenix refinements. | ||||||
| Refinement | Space: REAL / Protocol: FLEXIBLE FIT / Target criteria: cross-correlation coefficient | ||||||
| Output model |  PDB-5vrf: |
 Movie
Movie Controller
Controller


 UCSF Chimera
UCSF Chimera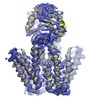








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)