登録情報 データベース : EMDB / ID : EMD-7335タイトル Cryo-EM structure of PRC2 bound to cofactors AEBP2 and JARID2 in the Extended Active State primary map 複合体 : Ternary complex of PRC2 with cofactors AEBP2 and JARID2タンパク質・ペプチド : Polycomb protein SUZ12タンパク質・ペプチド : Protein Jumonjiタンパク質・ペプチド : Protein Jumonjiタンパク質・ペプチド : Histone-lysine N-methyltransferase EZH2タンパク質・ペプチド : Polycomb protein EEDタンパク質・ペプチド : Histone-binding protein RBBP4タンパク質・ペプチド : JARID2-substrateタンパク質・ペプチド : Zinc finger protein AEBP2タンパク質・ペプチド : SUZ12機能・相同性 分子機能 ドメイン・相同性 構成要素
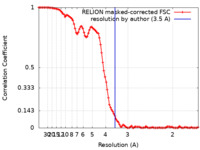
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.5 Å Kasinath V / Faini M / Poepsel S / Reif D / Feng A / Stjepanovic G / Aebersold R / Nogales E 資金援助 Organization Grant number 国 Howard Hughes Medical Institute (HHMI) European Research Council (ERC) 670821 European Union European Research Council (ERC) 233226 European Union
ジャーナル : Science / 年 : 2018タイトル : Structures of human PRC2 with its cofactors AEBP2 and JARID2.著者 : Vignesh Kasinath / Marco Faini / Simon Poepsel / Dvir Reif / Xinyu Ashlee Feng / Goran Stjepanovic / Ruedi Aebersold / Eva Nogales / 要旨 : Transcriptionally repressive histone H3 lysine 27 methylation by Polycomb repressive complex 2 (PRC2) is essential for cellular differentiation and development. Here we report cryo-electron ... Transcriptionally repressive histone H3 lysine 27 methylation by Polycomb repressive complex 2 (PRC2) is essential for cellular differentiation and development. Here we report cryo-electron microscopy structures of human PRC2 in a basal state and two distinct active states while in complex with its cofactors JARID2 and AEBP2. Both cofactors mimic the binding of histone H3 tails. JARID2, methylated by PRC2, mimics a methylated H3 tail to stimulate PRC2 activity, whereas AEBP2 interacts with the RBAP48 subunit, mimicking an unmodified H3 tail. SUZ12 interacts with all other subunits within the assembly and thus contributes to the stability of the complex. Our analysis defines the complete architecture of a functionally relevant PRC2 and provides a structural framework to understand its regulation by cofactors, histone tails, and RNA. 履歴 登録 2018年1月6日 - ヘッダ(付随情報) 公開 2018年1月24日 - マップ公開 2018年1月24日 - 更新 2020年8月12日 - 現状 2020年8月12日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 米国, European Union, 3件
米国, European Union, 3件  引用
引用 ジャーナル: Science / 年: 2018
ジャーナル: Science / 年: 2018

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_7335.map.gz
emd_7335.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-7335-v30.xml
emd-7335-v30.xml emd-7335.xml
emd-7335.xml EMDBヘッダ
EMDBヘッダ emd_7335_fsc.xml
emd_7335_fsc.xml FSCデータファイル
FSCデータファイル emd_7335.png
emd_7335.png emd_7335_half_map_1.map.gz
emd_7335_half_map_1.map.gz emd_7335_half_map_2.map.gz
emd_7335_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-7335
http://ftp.pdbj.org/pub/emdb/structures/EMD-7335 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7335
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7335 emd_7335_validation.pdf.gz
emd_7335_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_7335_full_validation.pdf.gz
emd_7335_full_validation.pdf.gz emd_7335_validation.xml.gz
emd_7335_validation.xml.gz emd_7335_validation.cif.gz
emd_7335_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7335
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7335 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7335
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7335 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_7335.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_7335.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー


































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)































 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)