+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6313 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 2.5A structure of lysozyme solved by MicroED | |||||||||
 マップデータ マップデータ | Lysozyme electron diffraction phased by molecular replacement. PDB ID 3J6K. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | lysozyme / microcrystal | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Lactose synthesis / Antimicrobial peptides / Neutrophil degranulation / beta-N-acetylglucosaminidase activity / cell wall macromolecule catabolic process / lysozyme / lysozyme activity / defense response to Gram-negative bacterium / killing of cells of another organism / defense response to Gram-positive bacterium ...Lactose synthesis / Antimicrobial peptides / Neutrophil degranulation / beta-N-acetylglucosaminidase activity / cell wall macromolecule catabolic process / lysozyme / lysozyme activity / defense response to Gram-negative bacterium / killing of cells of another organism / defense response to Gram-positive bacterium / defense response to bacterium / endoplasmic reticulum / extracellular space / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Nannenga BL / Shi D / Leslie AGW / Gonen T | |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2014 ジャーナル: Nat Methods / 年: 2014タイトル: High-resolution structure determination by continuous-rotation data collection in MicroED. 著者: Brent L Nannenga / Dan Shi / Andrew G W Leslie / Tamir Gonen /   要旨: MicroED uses very small three-dimensional protein crystals and electron diffraction for structure determination. We present an improved data collection protocol for MicroED called 'continuous ...MicroED uses very small three-dimensional protein crystals and electron diffraction for structure determination. We present an improved data collection protocol for MicroED called 'continuous rotation'. Microcrystals are continuously rotated during data collection, yielding more accurate data. The method enables data processing with the crystallographic software tool MOSFLM, which resulted in improved resolution for the model protein lysozyme. These improvements are paving the way for the broad implementation and application of MicroED in structural biology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6313.map.gz emd_6313.map.gz | 3.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6313-v30.xml emd-6313-v30.xml emd-6313.xml emd-6313.xml | 12 KB 12 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6313.gif 400_6313.gif 80_6313.gif 80_6313.gif | 59.8 KB 4.2 KB | ||
| マスクデータ |  emd_6313_msk_1.map emd_6313_msk_1.map | 965.4 KB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6313 http://ftp.pdbj.org/pub/emdb/structures/EMD-6313 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6313 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6313 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6313_validation.pdf.gz emd_6313_validation.pdf.gz | 412.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6313_full_validation.pdf.gz emd_6313_full_validation.pdf.gz | 412.2 KB | 表示 | |
| XML形式データ |  emd_6313_validation.xml.gz emd_6313_validation.xml.gz | 4.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6313 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6313 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6313 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6313 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3j6kMC  6342C  5a3eC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6313.map.gz / 形式: CCP4 / 大きさ: 3.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6313.map.gz / 形式: CCP4 / 大きさ: 3.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Lysozyme electron diffraction phased by molecular replacement. PDB ID 3J6K. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.60768 Å / Y: 0.60768 Å / Z: 0.62033 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
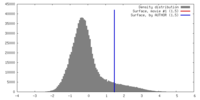
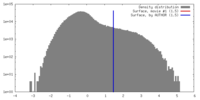
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 96 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-セグメンテーションマップ: This mask represents the map masked around the...
| 注釈 | This mask represents the map masked around the asymmetric unit (PDB 3J6K) | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_6313_msk_1.map emd_6313_msk_1.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
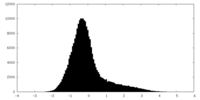
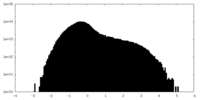
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hen egg white lysozyme
| 全体 | 名称: Hen egg white lysozyme |
|---|---|
| 要素 |
|
-超分子 #1000: Hen egg white lysozyme
| 超分子 | 名称: Hen egg white lysozyme / タイプ: sample / ID: 1000 / 詳細: lysozyme microcrystals / 集合状態: 1 / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 14.3 KDa |
-分子 #1: Lysozyme C
| 分子 | 名称: Lysozyme C / タイプ: protein_or_peptide / ID: 1 / Name.synonym: lysozyme / 詳細: lysozyme microcrystals / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.3 KDa |
| 配列 | UniProtKB: Lysozyme C |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線結晶学 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 200 mg/mL |
|---|---|
| 緩衝液 | pH: 4.5 詳細: 50 mM sodium acetate, 3.5 M sodium chloride, 15% PEG5000 |
| グリッド | 詳細: 300 mesh copper grid with holey carbon support |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 100 K / 装置: FEI VITROBOT MARK IV 手法: Add crystals to grid for 30 seconds and blot for 10-12 seconds. |
| 詳細 | batch crystallization |
| 結晶化 | 詳細: batch crystallization |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 90 K / 最高: 110 K / 平均: 100 K |
| 日付 | 2014年2月4日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 平均電子線量: 0.1 e/Å2 / カメラ長: 1500 詳細: Raw diffraction images are available upon request. Contact the Gonen lab for access. |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: DIFFRACTION |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt angle min: -45 / Tilt angle max: 45 / Tilt series - Axis1 - Min angle: -45 ° / Tilt series - Axis1 - Max angle: 45 ° |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.5 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES |
|---|---|
| 結晶パラメータ | 単位格子 - A: 75.96 Å / 単位格子 - B: 75.96 Å / 単位格子 - C: 37.22 Å / 単位格子 - γ: 90 ° / 単位格子 - α: 90 ° / 単位格子 - β: 90 ° / 空間群: P 43 21 2 |
 ムービー
ムービー コントローラー
コントローラー









 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)