+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4780 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Heterodimeric ABC exporter TmrAB under turnover conditions in asymmetric unlocked return conformation with wider opened intracellular gate | ||||||||||||
 マップデータ マップデータ | TmrAB in asymmetric unlocked return state under turnover conditions with wider opening of intracellular gate | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | ATP-binding cassette transporter / membrane protein / heterodimer / exporter / TRANSPORT PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ABC-type oligopeptide transporter activity / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Thermus thermophilus (バクテリア) / Thermus thermophilus (バクテリア) /  | ||||||||||||
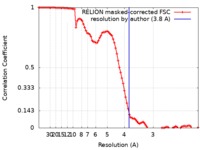
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | ||||||||||||
 データ登録者 データ登録者 | Januliene D / Hofmann S / Medhdipour AR / Thomas C / Hummer G / Tampe R / Moeller A | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Conformation space of a heterodimeric ABC exporter under turnover conditions. 著者: Susanne Hofmann / Dovile Januliene / Ahmad R Mehdipour / Christoph Thomas / Erich Stefan / Stefan Brüchert / Benedikt T Kuhn / Eric R Geertsma / Gerhard Hummer / Robert Tampé / Arne Moeller /  要旨: Cryo-electron microscopy (cryo-EM) has the capacity to capture molecular machines in action. ATP-binding cassette (ABC) exporters are highly dynamic membrane proteins that extrude a wide range of ...Cryo-electron microscopy (cryo-EM) has the capacity to capture molecular machines in action. ATP-binding cassette (ABC) exporters are highly dynamic membrane proteins that extrude a wide range of substances from the cytosol and thereby contribute to essential cellular processes, adaptive immunity and multidrug resistance. Despite their importance, the coupling of nucleotide binding, hydrolysis and release to the conformational dynamics of these proteins remains poorly resolved, especially for heterodimeric and/or asymmetric ABC exporters that are abundant in humans. Here we present eight high-resolution cryo-EM structures that delineate the full functional cycle of an asymmetric ABC exporter in a lipid environment. Cryo-EM analysis under active turnover conditions reveals distinct inward-facing (IF) conformations-one of them with a bound peptide substrate-and previously undescribed asymmetric post-hydrolysis states with dimerized nucleotide-binding domains and a closed extracellular gate. By decreasing the rate of ATP hydrolysis, we could capture an outward-facing (OF) open conformation-an otherwise transient state vulnerable to substrate re-entry. The ATP-bound pre-hydrolysis and vanadate-trapped states are conformationally equivalent; both comprise co-existing OF conformations with open and closed extracellular gates. By contrast, the post-hydrolysis states from the turnover experiment exhibit asymmetric ATP and ADP occlusion after phosphate release from the canonical site and display a progressive separation of the nucleotide-binding domains and unlocking of the intracellular gate. Our findings reveal that phosphate release, not ATP hydrolysis, triggers the return of the exporter to the IF conformation. By mapping the conformational landscape during active turnover, aided by mutational and chemical modulation of kinetic rates to trap the key intermediates, we resolved fundamental steps of the substrate translocation cycle of asymmetric ABC transporters. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4780.map.gz emd_4780.map.gz | 3.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4780-v30.xml emd-4780-v30.xml emd-4780.xml emd-4780.xml | 26.9 KB 26.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4780_fsc.xml emd_4780_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4780.png emd_4780.png | 41.4 KB | ||
| マスクデータ |  emd_4780_msk_1.map emd_4780_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-4780.cif.gz emd-4780.cif.gz | 7.9 KB | ||
| その他 |  emd_4780_half_map_1.map.gz emd_4780_half_map_1.map.gz emd_4780_half_map_2.map.gz emd_4780_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4780 http://ftp.pdbj.org/pub/emdb/structures/EMD-4780 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4780 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4780 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4780_validation.pdf.gz emd_4780_validation.pdf.gz | 1008.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4780_full_validation.pdf.gz emd_4780_full_validation.pdf.gz | 1007.7 KB | 表示 | |
| XML形式データ |  emd_4780_validation.xml.gz emd_4780_validation.xml.gz | 16.3 KB | 表示 | |
| CIF形式データ |  emd_4780_validation.cif.gz emd_4780_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4780 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4780 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4780 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4780 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ramMC  4773C  4774C  4775C  4776C  4777C  4778C  4779C  4781C  6rafC  6ragC  6rahC  6raiC  6rajC  6rakC  6ralC  6ranC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4780.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4780.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TmrAB in asymmetric unlocked return state under turnover conditions with wider opening of intracellular gate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.077 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4780_msk_1.map emd_4780_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_4780_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_4780_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TmrAB under turnover conditions in asymmetric unlocked return con...
| 全体 | 名称: TmrAB under turnover conditions in asymmetric unlocked return conformation with wider opened intracellular gate |
|---|---|
| 要素 |
|
-超分子 #1: TmrAB under turnover conditions in asymmetric unlocked return con...
| 超分子 | 名称: TmrAB under turnover conditions in asymmetric unlocked return conformation with wider opened intracellular gate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 150 KDa |
-超分子 #2: TmrAB
| 超分子 | 名称: TmrAB / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
-超分子 #3: nanobody
| 超分子 | 名称: nanobody / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Multidrug resistance ABC transporter ATP-binding and permease protein
| 分子 | 名称: Multidrug resistance ABC transporter ATP-binding and permease protein タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
| 分子量 | 理論値: 70.664797 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTEDTYSKAF DRALFARILR YVWPYRLQVV LALLFLLVVT LAAAATPLFF KWAIDLALVP TEPRPLAERF HLLLWISLGF LAVRAVHFA ATYGETYLIQ WVGQRVLFDL RSDLFAKLMR LHPGFYDRNP VGRLMTRVTS DVDAINQFIT GGLVGVIADL F TLVGLLGF ...文字列: MTEDTYSKAF DRALFARILR YVWPYRLQVV LALLFLLVVT LAAAATPLFF KWAIDLALVP TEPRPLAERF HLLLWISLGF LAVRAVHFA ATYGETYLIQ WVGQRVLFDL RSDLFAKLMR LHPGFYDRNP VGRLMTRVTS DVDAINQFIT GGLVGVIADL F TLVGLLGF MLFLSPKLTL VVLLVAPVLL AVTTWVRLGM RSAYREMRLR LARVNAALQE NLSGVETIQL FVKEREREEK FD RLNRDLF RAWVEIIRWF ALFFPVVGFL GDFAVASLVY YGGGEVVRGA VSLGLLVAFV DYTRQLFQPL QDLSDKFNLF QGA MASAER IFGVLDTEEE LKDPEDPTPI RGFRGEVEFR DVWLAYTPKG VEPTEKDWVL KGVSFRVRPG EKVALVGATG AGKT SVVSL IARFYDPQRG CVFLDGVDVR RYRQEELRRH VGIVLQEPFL FSGTVLDNLR LFDPSVPPER VEEVARFLGA HEFIL RLPK GYQTVLGERG AGLSTGEKQL LALVRALLAS PDILLILDEA TASVDSETEK RLQEALYKAM EGRTSLIIAH RLSTIR HVD RILVFRKGRL VEEGSHEELL AKGGYYAALY RLQFQEAKLG GGGENLYFQG HHHHHHHHHH UniProtKB: Multidrug resistance ABC transporter ATP-binding and permease protein |
-分子 #2: Multidrug resistance ABC transporter ATP-binding and permease protein
| 分子 | 名称: Multidrug resistance ABC transporter ATP-binding and permease protein タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
| 分子量 | 理論値: 64.634457 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTGRSAAPLL RRLWPYVGRY RWRYLWAVLA GLVSIFFFVL TPYFLRLAVD AVQAGRGFGV YALAIVASAA LSGLLSYAMR RLAVVASRQ VEYDLRRDLL HHLLTLDRDF YHKHRVGDLM NRLNTDLSAV REMVGPGILM GSRLSFLVLL AFLSMYAVNA R LAFYLTLI ...文字列: MTGRSAAPLL RRLWPYVGRY RWRYLWAVLA GLVSIFFFVL TPYFLRLAVD AVQAGRGFGV YALAIVASAA LSGLLSYAMR RLAVVASRQ VEYDLRRDLL HHLLTLDRDF YHKHRVGDLM NRLNTDLSAV REMVGPGILM GSRLSFLVLL AFLSMYAVNA R LAFYLTLI LPGIFLAMRF LLRLIDRRYR EAQEVFDRIS TLAQEAFSGI RVVKGYALER RMVAWFQDLN RLYVEKSLAL AR VEGPLHA LLGFLMGFAF LTVLWAGGAM VVRGELSVGE LVQFNAYLAQ LTWPILGLGW VMALYQRGLT SLRRLFELLD EKP AIRDED PLPLALEDLS GEVRFEGVGL KRDGRWLLRG LTLTIPEGMT LGITGRTGSG KSLLAALVPR LLDPSEGRVY VGGH EARRI PLAVLRKAVG VAPQEPFLFS ETILENIAFG LDEVDRERVE WAARLAGIHE EILAFPKGYE TVLGERGITL SGGQR QRVA LARALAKRPK ILILDDALSA VDAETEARIL QGLKTVLGKQ TTLLISHRTA ALRHADWIIV LDGGRIVEEG THESLL QAG GLYAEMDRLQ KEVEA UniProtKB: Multidrug resistance ABC transporter ATP-binding and permease protein |
-分子 #3: Nanobody Nb9F10
| 分子 | 名称: Nanobody Nb9F10 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.594405 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAQLQLVESG GGLVQPGDSL RLSCAVSGSA LDYNAIGWFR QAPGKEREGV ACISKITGNT AYADSVKGRF TISRDNAKNT VHLQMNSLK PEDTAVYYCA TVTAVLLPGR CVPGKYWGQG TPVTVSSHHH HHHEPEA |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil, UltrAuFoil / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均露光時間: 8.0 sec. / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)