+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4460 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Salmonella AcrB solubilised in the SMA copolymer | |||||||||
 マップデータ マップデータ | Local resolution filtered map of Salmonella AcrB | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報xenobiotic transport / efflux transmembrane transporter activity / membrane => GO:0016020 / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Salmonella (サルモネラ菌) Salmonella (サルモネラ菌) | |||||||||
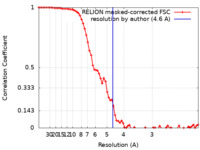
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Johnson RM / Bavro VN / Postis V / Muench SP | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Microorganisms / 年: 2020 ジャーナル: Microorganisms / 年: 2020タイトル: Cryo-EM Structure and Molecular Dynamics Analysis of the Fluoroquinolone Resistant Mutant of the AcrB Transporter from . 著者: Rachel M Johnson / Chiara Fais / Mayuriben Parmar / Harish Cheruvara / Robert L Marshall / Sophie J Hesketh / Matthew C Feasey / Paolo Ruggerone / Attilio V Vargiu / Vincent L G Postis / ...著者: Rachel M Johnson / Chiara Fais / Mayuriben Parmar / Harish Cheruvara / Robert L Marshall / Sophie J Hesketh / Matthew C Feasey / Paolo Ruggerone / Attilio V Vargiu / Vincent L G Postis / Stephen P Muench / Vassiliy N Bavro /   要旨: is an important genus of Gram-negative pathogens, treatment of which has become problematic due to increases in antimicrobial resistance. This is partly attributable to the overexpression of ... is an important genus of Gram-negative pathogens, treatment of which has become problematic due to increases in antimicrobial resistance. This is partly attributable to the overexpression of tripartite efflux pumps, particularly the constitutively expressed AcrAB-TolC. Despite its clinical importance, the structure of the AcrB transporter remained unknown to-date, with much of our structural understanding coming from the orthologue. Here, by taking advantage of the styrene maleic acid (SMA) technology to isolate membrane proteins with closely associated lipids, we report the very first experimental structure of AcrB transporter. Furthermore, this novel structure provides additional insight into mechanisms of drug efflux as it bears the mutation (G288D), originating from a clinical isolate of Typhimurium presenting an increased resistance to fluoroquinolones. Experimental data are complemented by state-of-the-art molecular dynamics (MD) simulations on both the wild type and G288D variant of AcrB. Together, these reveal several important differences with respect to the protein, providing insights into the role of the G288D mutation in increasing drug efflux and extending our understanding of the mechanisms underlying antibiotic resistance. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4460.map.gz emd_4460.map.gz | 20.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4460-v30.xml emd-4460-v30.xml emd-4460.xml emd-4460.xml | 24.1 KB 24.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4460_fsc.xml emd_4460_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4460.png emd_4460.png | 156.6 KB | ||
| その他 |  emd_4460_additional_1.map.gz emd_4460_additional_1.map.gz emd_4460_additional_2.map.gz emd_4460_additional_2.map.gz emd_4460_half_map_1.map.gz emd_4460_half_map_1.map.gz emd_4460_half_map_2.map.gz emd_4460_half_map_2.map.gz | 6.8 MB 23.2 MB 23.3 MB 23.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4460 http://ftp.pdbj.org/pub/emdb/structures/EMD-4460 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4460 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4460 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4460_validation.pdf.gz emd_4460_validation.pdf.gz | 361 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4460_full_validation.pdf.gz emd_4460_full_validation.pdf.gz | 360.1 KB | 表示 | |
| XML形式データ |  emd_4460_validation.xml.gz emd_4460_validation.xml.gz | 13.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4460 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4460 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4460 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4460 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4460.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4460.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution filtered map of Salmonella AcrB | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Post-processed (sharpened) cryo-EM map of Salmonella AcrB
| ファイル | emd_4460_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed (sharpened) cryo-EM map of Salmonella AcrB | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
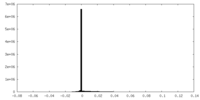
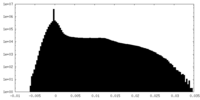
| 密度ヒストグラム |
-追加マップ: Unsharpened cryo-EM map of Salmonella AcrB
| ファイル | emd_4460_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened cryo-EM map of Salmonella AcrB | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
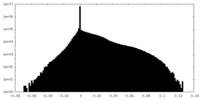
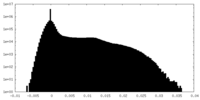
| 密度ヒストグラム |
-ハーフマップ: Half map 1 for Salmonella AcrB
| ファイル | emd_4460_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 for Salmonella AcrB | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
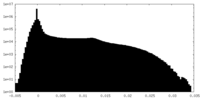
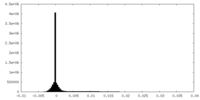
| 密度ヒストグラム |
-ハーフマップ: Half map 2 for Salmonella AcrB
| ファイル | emd_4460_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 for Salmonella AcrB | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
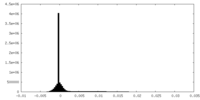
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Salmonella AcrB
| 全体 | 名称: Salmonella AcrB |
|---|---|
| 要素 |
|
-超分子 #1: Salmonella AcrB
| 超分子 | 名称: Salmonella AcrB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Cryo-EM structure of Salmonella multidrug bacterial efflux pump AcrB solubilised in SMA co-polymer |
|---|---|
| 由来(天然) | 生物種:  Salmonella (サルモネラ菌) Salmonella (サルモネラ菌) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 340 KDa |
-分子 #1: Salmonella multidrug efflux transporter AcrB in a SMALP platform
| 分子 | 名称: Salmonella multidrug efflux transporter AcrB in a SMALP platform タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Salmonella (サルモネラ菌) Salmonella (サルモネラ菌) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPNFFIDRPI FAWVIAIIIM LAGGLAILKL PVAQYPTIAP PAVTISATYP GADAKTVQDT VTQVIEQNM NGIDNLMYMS SNSDSTGTVQ ITLTFESGTD ADIAQVQVQN KLQLAMPLLP Q EVQQQGVS VEKSSSSFLM VVGVINTDGT MTQEDISDYV AANMKDPISR ...文字列: MPNFFIDRPI FAWVIAIIIM LAGGLAILKL PVAQYPTIAP PAVTISATYP GADAKTVQDT VTQVIEQNM NGIDNLMYMS SNSDSTGTVQ ITLTFESGTD ADIAQVQVQN KLQLAMPLLP Q EVQQQGVS VEKSSSSFLM VVGVINTDGT MTQEDISDYV AANMKDPISR TSGVGDVQLF GS QYAMRIW MNPTELTKYQ LTPVDVINAI KAQNAQVAAG QLGGTPPVKG QQLNASIIAQ TRL TSTDEF GKILLKVNQD GSQVRLRDVA KIELGGENYD VIAKFNGQPA SGLGIKLATG ANAL DTATA IRAELKKMEP FFPPGMKIVY PYDTTPFVKI SIHEVVKTLV EAIILVFLVM YLFLQ NFRA TLIPTIAVPV VLLGTFAVLA AFGFSINTLT MFGMVLAIGL LVDDAIVVVE NVERVM TEE GLPPKEATRK SMGQIQGALV GIAMVLSAVF IPMAFFGGST GAIYRQFSIT IVSAMAL SV LVALILTPAL CATMLKPVAK GDHGEGKKGF FGWFNRLFDK STHHYTDSVG NILRSTGR Y LLLYLIIVVG MAYLFVRLPS SFLPDEDQGV FLTMVQLPAG ATQERTQKVL DEVTDYYLN KEKANVESVF AVNGFGFAGR GQNTGIAFVS LKDWADRPGE KNKVEAITQR ATAAFSQIKD AMVFAFNLP AIVELGTATG FDFELIDQAG LGHEKLTQAR NQLFGEVAKY PDLLVGVRPN G LEDTPQFK IDIDQEKAQA LGVSISDINT TLGAAWGGSY VNDFIDRGRV KKVYVMSEAK YR MLPDDIN DWYVRGSDGQ MVPFSAFSSS RWEYGSPRLE RYNGLPSMEI LGQAAPGKST GEA MAMMEE LASKLPSGIG YDWTGMSYQE RLSGNQAPAL YAISLIVVFL CLAALYESWS IPFS VMLVV PLGVIGALLA ATFRGLTNDV YFQVGLLTTI GLSAKNAILI VEFAKDLMDK EGKGL VEAT LEAVRMRLRP ILMTSLAFML GVMPLVISSG AGSGAQNAVG TGVLGGMVTA TVLAIF FVP VFFVVVRRRF SRKSEDIEHS HSTEHR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Salmonella AcrB was prepared in buffer containing 50mM Tris (pH8) with 500 mM Nacl and 10% glycerol. For EM grids, the sample was diluted into buffer containing 50 mM Tris and 150 mM NaCl. | |||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Grids were prepared using a Vitrobot Mark IV and blotted with Ash-free Whatman filter paper (No. 50) for 6 seconds using a blot force of 6.. | |||||||||
| 詳細 | Salmonella AcrB was prepared in buffer containing 50mM Tris (pH8) with 500 mM Nacl and 10% glycerol. For EM grids, the sample was diluted into buffer containing 50 mM Tris and 150 mM NaCl. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 3210 / 平均露光時間: 10.0 sec. / 平均電子線量: 61.7 e/Å2 / 詳細: 3,210 images were collected in a 72 hour period. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.0045 µm / 最小 デフォーカス(公称値): 0.0015 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X