+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Dia1 at the Barbed End of F-Actin | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Actin / Filament / Elongation / Ends / CYTOSOLIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of neuron projection regeneration / multicellular organismal locomotion / RHOF GTPase cycle / RHOB GTPase cycle / ERBB2 Regulates Cell Motility / RHOC GTPase cycle / RHOD GTPase cycle / RHOA GTPase cycle / actin nucleation / neuron projection retraction ...negative regulation of neuron projection regeneration / multicellular organismal locomotion / RHOF GTPase cycle / RHOB GTPase cycle / ERBB2 Regulates Cell Motility / RHOC GTPase cycle / RHOD GTPase cycle / RHOA GTPase cycle / actin nucleation / neuron projection retraction / RHO GTPases Activate Formins / profilin binding / regulation of microtubule-based process / axon midline choice point recognition / cytoskeletal motor activator activity / tropomyosin binding / mesenchyme migration / troponin I binding / myosin heavy chain binding / filamentous actin / actin filament bundle / brush border / skeletal muscle thin filament assembly / striated muscle thin filament / actin filament bundle assembly / synaptic vesicle endocytosis / skeletal muscle myofibril / actin monomer binding / ephrin receptor signaling pathway / skeletal muscle fiber development / stress fiber / titin binding / cytoskeleton organization / Neutrophil degranulation / actin filament polymerization / filopodium / actin filament / sensory perception of sound / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / protein localization / brain development / small GTPase binding / ruffle membrane / spindle / calcium-dependent protein binding / neuron projection development / presynapse / lamellipodium / actin binding / gene expression / regulation of cell shape / cell body / actin cytoskeleton organization / transmembrane transporter binding / hydrolase activity / positive regulation of cell migration / neuron projection / protein domain specific binding / centrosome / calcium ion binding / positive regulation of gene expression / magnesium ion binding / ATP binding / identical protein binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
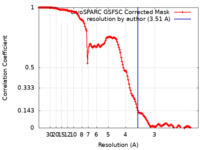
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.51 Å | |||||||||
 データ登録者 データ登録者 | Palmer NJ / Barrie KR / Dominguez R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Mechanisms of actin filament severing and elongation by formins. 著者: Nicholas J Palmer / Kyle R Barrie / Roberto Dominguez /  要旨: Humans express fifteen formins, playing crucial roles in actin-based processes, such as cytokinesis, cell motility, and mechanotransduction . However, the lack of structures bound to the actin ...Humans express fifteen formins, playing crucial roles in actin-based processes, such as cytokinesis, cell motility, and mechanotransduction . However, the lack of structures bound to the actin filament (F-actin) has been a major impediment to understanding formin function. While formins are known for their ability to nucleate and elongate F-actin , some formins can additionally depolymerize, sever, or bundle F-actin. Two mammalian formins, inverted formin-2 (INF2) and diaphanous-1 (Dia1), exemplify this diversity. INF2 displays potent severing activity but elongates weakly , whereas Dia1 has potent elongation activity but does not sever . Using cryo-electron microscopy (cryo-EM), we reveal five structural states of INF2 and two of Dia1 bound to the middle and barbed end of F-actin. INF2 and Dia1 bind differently to these sites, consistent with their distinct activities. The FH2 and WH2 domains of INF2 are positioned to sever F-actin, whereas Dia1 appears unsuited for severing. Structures also show how profilin-actin is delivered to the fast-growing barbed end, and how this is followed by a transition of the incoming monomer into the F-actin conformation and the release of profilin. Combined, the seven structures presented here provide step-by-step visualization of the mechanisms of F-actin severing and elongation by formins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44099.map.gz emd_44099.map.gz | 133.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44099-v30.xml emd-44099-v30.xml emd-44099.xml emd-44099.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44099_fsc.xml emd_44099_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44099.png emd_44099.png | 23.6 KB | ||
| マスクデータ |  emd_44099_msk_1.map emd_44099_msk_1.map | 266.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-44099.cif.gz emd-44099.cif.gz | 6.6 KB | ||
| その他 |  emd_44099_half_map_1.map.gz emd_44099_half_map_1.map.gz emd_44099_half_map_2.map.gz emd_44099_half_map_2.map.gz | 247.8 MB 247.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44099 http://ftp.pdbj.org/pub/emdb/structures/EMD-44099 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44099 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44099 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44099_validation.pdf.gz emd_44099_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44099_full_validation.pdf.gz emd_44099_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_44099_validation.xml.gz emd_44099_validation.xml.gz | 22.6 KB | 表示 | |
| CIF形式データ |  emd_44099_validation.cif.gz emd_44099_validation.cif.gz | 29.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44099 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44099 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44099 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44099 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9b27MC  9az4C  9azpC  9azqC  9b03C  9b0kC  9b3dC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44099.map.gz / 形式: CCP4 / 大きさ: 266.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44099.map.gz / 形式: CCP4 / 大きさ: 266.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
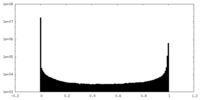
-マスク #1
| ファイル |  emd_44099_msk_1.map emd_44099_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
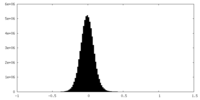
-ハーフマップ: #2
| ファイル | emd_44099_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
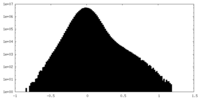
-ハーフマップ: #1
| ファイル | emd_44099_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of mDia1 dimer bound at the barbed end of actin filaments.
| 全体 | 名称: Structure of mDia1 dimer bound at the barbed end of actin filaments. |
|---|---|
| 要素 |
|
-超分子 #1: Structure of mDia1 dimer bound at the barbed end of actin filaments.
| 超分子 | 名称: Structure of mDia1 dimer bound at the barbed end of actin filaments. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 252 KDa |
-超分子 #2: mDia1 dimer
| 超分子 | 名称: mDia1 dimer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Actin filament
| 超分子 | 名称: Actin filament / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Actin, alpha skeletal muscle
| 分子 | 名称: Actin, alpha skeletal muscle / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.387227 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TTALVCDNGS GLVKAGFAGD DAPRAVFPSI VGRPRHQGVM VGMGQKDSYV GDEAQSKRGI LTLKYPIE(HIC)G IITNWD DME KIWHHTFYNE LRVAPEEHPT LLTEAPLNPK ANREKMTQIM FETFNVPAMY VAIQAVLSLY ASGRTTGIVL DSGDGVT HN ...文字列: TTALVCDNGS GLVKAGFAGD DAPRAVFPSI VGRPRHQGVM VGMGQKDSYV GDEAQSKRGI LTLKYPIE(HIC)G IITNWD DME KIWHHTFYNE LRVAPEEHPT LLTEAPLNPK ANREKMTQIM FETFNVPAMY VAIQAVLSLY ASGRTTGIVL DSGDGVT HN VPIYEGYALP HAIMRLDLAG RDLTDYLMKI LTERGYSFVT TAEREIVRDI KEKLCYVALD FENEMATAAS SSSLEKSY E LPDGQVITIG NERFRCPETL FQPSFIGMES AGIHETTYNS IMKCDIDIRK DLYANNVMSG GTTMYPGIAD RMQKEITAL APSTMKIKII APPERKYSVW IGGSILASLS TFQQMWITKQ EYDEAGPSIV HRKCF UniProtKB: Actin, alpha skeletal muscle |
-分子 #2: Protein diaphanous homolog 1
| 分子 | 名称: Protein diaphanous homolog 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.189508 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VLPFGLTPKK VYKPEVQLRR PNWSKFVAED LSQDCFWTKV KEDRFENNEL FAKLTLAFSA QTKTSKAKKD QEGGEEKKSV QKKKVKELK VLDSKTAQNL SIFLGSFRMP YQEIKNVILE VNEAVLTESM IQNLIKQMPE PEQLKMLSEL KEEYDDLAES E QFGVVMGT ...文字列: VLPFGLTPKK VYKPEVQLRR PNWSKFVAED LSQDCFWTKV KEDRFENNEL FAKLTLAFSA QTKTSKAKKD QEGGEEKKSV QKKKVKELK VLDSKTAQNL SIFLGSFRMP YQEIKNVILE VNEAVLTESM IQNLIKQMPE PEQLKMLSEL KEEYDDLAES E QFGVVMGT VPRLRPRLNA ILFKLQFSEQ VENIKPEIVS VTAACEELRK SENFSSLLEL TLLVGNYMNA GSRNAGAFGF NI SFLCKLR DTKSADQKMT LLHFLAELCE NDHPEVLKFP DELAHVEKAS RVSAENLQKS LDQMKKQIAD VERDVQNFPA ATD EKDKFV EKMTSFVKDA QEQYNKLRMM HSNMETLYKE LGDYFVFDPK KLSVEEFFMD LHNFRNMFLQ AVKENQKRRE TEEK MRRAK LAKEKAEKER LEKQQKR UniProtKB: Protein diaphanous homolog 1 |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 6 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.48 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 100 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)