+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Myxococcus xanthus EncA encapsulin shell loaded with EncD cargo | |||||||||
 マップデータ マップデータ | EncA-EncD encapsulin cryo-EM map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Encapsulin / EncA / EncD / flavin / flavin mononucleotide / iron / ferroxidase / ferritin / ferric reductase / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 | Type 1 encapsulin shell protein / Encapsulating protein for peroxidase / encapsulin nanocompartment / iron ion transport / intracellular iron ion homeostasis / metal ion binding / Type 1 encapsulin shell protein EncA / Encapsulin nanocompartment cargo protein EncD 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Myxococcus xanthus (バクテリア) Myxococcus xanthus (バクテリア) | |||||||||
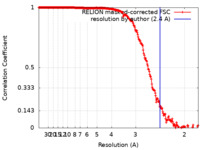
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Eren E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: encapsulin cargo protein EncD is a flavin-binding protein with ferric reductase activity. 著者: Elif Eren / Norman R Watts / James F Conway / Paul T Wingfield /  要旨: Encapsulins are protein nanocompartments that regulate cellular metabolism in several bacteria and archaea. encapsulins protect the bacterial cells against oxidative stress by sequestering cytosolic ...Encapsulins are protein nanocompartments that regulate cellular metabolism in several bacteria and archaea. encapsulins protect the bacterial cells against oxidative stress by sequestering cytosolic iron. These encapsulins are formed by the shell protein EncA and three cargo proteins: EncB, EncC, and EncD. EncB and EncC form rotationally symmetric decamers with ferroxidase centers (FOCs) that oxidize Fe to Fe for iron storage in mineral form. However, the structure and function of the third cargo protein, EncD, have yet to be determined. Here, we report the x-ray crystal structure of EncD in complex with flavin mononucleotide. EncD forms an α-helical hairpin arranged as an antiparallel dimer, but unlike other flavin-binding proteins, it has no β-sheet, showing that EncD and its homologs represent a unique class of bacterial flavin-binding proteins. The cryo-EM structure of EncA-EncD encapsulins confirms that EncD binds to the interior of the EncA shell via its C-terminal targeting peptide. With only 100 amino acids, the EncD α-helical dimer forms the smallest flavin-binding domain observed to date. Unlike EncB and EncC, EncD lacks a FOC, and our biochemical results show that EncD instead is a NAD(P)H-dependent ferric reductase, indicating that the encapsulins act as an integrated system for iron homeostasis. Overall, this work contributes to our understanding of bacterial metabolism and could lead to the development of technologies for iron biomineralization and the production of iron-containing materials for the treatment of various diseases associated with oxidative stress. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43290.map.gz emd_43290.map.gz | 73.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43290-v30.xml emd-43290-v30.xml emd-43290.xml emd-43290.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43290_fsc.xml emd_43290_fsc.xml | 22.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43290.png emd_43290.png | 255.8 KB | ||
| Filedesc metadata |  emd-43290.cif.gz emd-43290.cif.gz | 5.7 KB | ||
| その他 |  emd_43290_half_map_1.map.gz emd_43290_half_map_1.map.gz emd_43290_half_map_2.map.gz emd_43290_half_map_2.map.gz | 471.3 MB 470.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43290 http://ftp.pdbj.org/pub/emdb/structures/EMD-43290 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43290 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43290 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43290_validation.pdf.gz emd_43290_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43290_full_validation.pdf.gz emd_43290_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_43290_validation.xml.gz emd_43290_validation.xml.gz | 28 KB | 表示 | |
| CIF形式データ |  emd_43290_validation.cif.gz emd_43290_validation.cif.gz | 37.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43290 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43290 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43290 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43290 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8vjoMC  8vjnC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43290.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43290.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EncA-EncD encapsulin cryo-EM map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.92 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: EncA-EncD halfmap1
| ファイル | emd_43290_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EncA-EncD halfmap1 | ||||||||||||
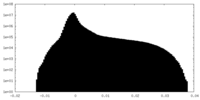
| 投影像・断面図 |
| ||||||||||||
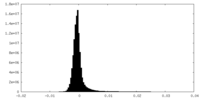
| 密度ヒストグラム |
-ハーフマップ: EncA-EncD halfmap2
| ファイル | emd_43290_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EncA-EncD halfmap2 | ||||||||||||
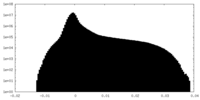
| 投影像・断面図 |
| ||||||||||||
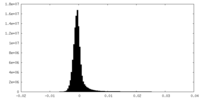
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Icosahedral encapsulin EncA particles in complex with cargo prote...
| 全体 | 名称: Icosahedral encapsulin EncA particles in complex with cargo protein EncD |
|---|---|
| 要素 |
|
-超分子 #1: Icosahedral encapsulin EncA particles in complex with cargo prote...
| 超分子 | 名称: Icosahedral encapsulin EncA particles in complex with cargo protein EncD タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Myxococcus xanthus (バクテリア) Myxococcus xanthus (バクテリア) |
-分子 #1: EncA
| 分子 | 名称: EncA / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Myxococcus xanthus (バクテリア) Myxococcus xanthus (バクテリア) |
| 分子量 | 理論値: 33.505074 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHMPL EPHFMPDFLG HAENPLREEE WARLNETVIQ VARRSLVGRR ILDIYGPLGA GVQTVPYDEF QGVSPGAVDI VGEQETAMV FTDARKFKTI PIIYKDFLLH WRDIEAARTH NMPLDVSAAA GAAALCAQQE DELIFYGDAR LGYEGLMTAN G RLTVPLGD ...文字列: MHHHHHHMPL EPHFMPDFLG HAENPLREEE WARLNETVIQ VARRSLVGRR ILDIYGPLGA GVQTVPYDEF QGVSPGAVDI VGEQETAMV FTDARKFKTI PIIYKDFLLH WRDIEAARTH NMPLDVSAAA GAAALCAQQE DELIFYGDAR LGYEGLMTAN G RLTVPLGD WTSPGGGFQA IVEATRKLNE QGHFGPYAVV LSPRLYSQLH RIYEKTGVLE IETIRQLASD GVYQSNRLRG ES GVVVSTG RENMDLAVSM DMVAAYLGAS RMNHPFRVLE ALLLRIKHPD AICTLEGAGA TERR UniProtKB: Type 1 encapsulin shell protein EncA |
-分子 #2: Encapsulin nanocompartment cargo protein EncD
| 分子 | 名称: Encapsulin nanocompartment cargo protein EncD / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Myxococcus xanthus (バクテリア) Myxococcus xanthus (バクテリア) |
| 分子量 | 理論値: 859.992 Da |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GLTVGSLRG UniProtKB: Encapsulin nanocompartment cargo protein EncD |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.3 / 詳細: 20mM HEPES, 150mM NaCl |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X