+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the E. coli clamp loader bound to the beta clamp in a Open-RNAp/t conformation | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacterial Clamp Loader Complex / REPLICATION / TRANSFERASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA polymerase III, clamp loader complex / Hda-beta clamp complex / bacterial-type DNA replication / replication inhibiting complex / DNA clamp loader activity / DNA polymerase III complex / replisome / regulation of DNA-templated DNA replication initiation / DNA strand elongation involved in DNA replication / DNA polymerase processivity factor activity ...DNA polymerase III, clamp loader complex / Hda-beta clamp complex / bacterial-type DNA replication / replication inhibiting complex / DNA clamp loader activity / DNA polymerase III complex / replisome / regulation of DNA-templated DNA replication initiation / DNA strand elongation involved in DNA replication / DNA polymerase processivity factor activity / error-prone translesion synthesis / negative regulation of DNA-templated DNA replication initiation / 3'-5' exonuclease activity / ribonucleoside triphosphate phosphatase activity / DNA-templated DNA replication / DNA replication / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / viral translational frameshifting / DNA damage response / protein homodimerization activity / ATP hydrolysis activity / DNA binding / ATP binding / identical protein binding / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
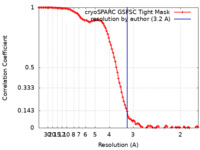
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Landeck JT / Kelch BA | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2024 ジャーナル: J Biol Chem / 年: 2024タイトル: Differences between bacteria and eukaryotes in clamp loader mechanism, a conserved process underlying DNA replication. 著者: Jacob T Landeck / Joshua Pajak / Emily K Norman / Emma L Sedivy / Brian A Kelch /  要旨: Clamp loaders are pentameric ATPases that place circular sliding clamps onto DNA, where they function in DNA replication and genome integrity. The central activity of a clamp loader is the opening of ...Clamp loaders are pentameric ATPases that place circular sliding clamps onto DNA, where they function in DNA replication and genome integrity. The central activity of a clamp loader is the opening of the ring-shaped sliding clamp and the subsequent binding to primer-template (p/t)-junctions. The general architecture of clamp loaders is conserved across all life, suggesting that their mechanism is retained. Recent structural studies of the eukaryotic clamp loader replication factor C (RFC) revealed that it functions using a crab-claw mechanism, where clamp opening is coupled to a massive conformational change in the loader. Here we investigate the clamp loading mechanism of the Escherichia coli clamp loader at high resolution using cryo-electron microscopy. We find that the E. coli clamp loader opens the clamp using a crab-claw motion at a single pivot point, whereas the eukaryotic RFC loader uses motions distributed across the complex. Furthermore, we find clamp opening occurs in multiple steps, starting with a partly open state with a spiral conformation, and proceeding to a wide open clamp in a surprising planar geometry. Finally, our structures in the presence of p/t-junctions illustrate how the clamp closes around p/t-junctions and how the clamp loader initiates release from the loaded clamp. Our results reveal mechanistic distinctions in a macromolecular machine that is conserved across all domains of life. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43102.map.gz emd_43102.map.gz | 97.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43102-v30.xml emd-43102-v30.xml emd-43102.xml emd-43102.xml | 23.6 KB 23.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43102_fsc.xml emd_43102_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43102.png emd_43102.png | 64.7 KB | ||
| Filedesc metadata |  emd-43102.cif.gz emd-43102.cif.gz | 7.7 KB | ||
| その他 |  emd_43102_half_map_1.map.gz emd_43102_half_map_1.map.gz emd_43102_half_map_2.map.gz emd_43102_half_map_2.map.gz | 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43102 http://ftp.pdbj.org/pub/emdb/structures/EMD-43102 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43102 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43102 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43102_validation.pdf.gz emd_43102_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43102_full_validation.pdf.gz emd_43102_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_43102_validation.xml.gz emd_43102_validation.xml.gz | 18.3 KB | 表示 | |
| CIF形式データ |  emd_43102_validation.cif.gz emd_43102_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43102 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43102 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43102 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43102 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8vatMC  8valC  8vamC  8vanC  8vapC  8vaqC  8varC  8vasC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43102.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43102.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.88 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_43102_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_43102_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Structure of the E. coli clamp loader bound to the beta clamp in ...
+超分子 #1: Structure of the E. coli clamp loader bound to the beta clamp in ...
+分子 #1: DNA polymerase III subunit delta
+分子 #2: DNA polymerase III subunit tau
+分子 #3: DNA polymerase III subunit delta'
+分子 #4: Beta sliding clamp
+分子 #5: RNA (5'-R(P*AP*GP*UP*GP*GP*UP*GP*UP*CP*UP*G)-3')
+分子 #6: DNA (5'-D(P*TP*TP*TP*TP*TP*TP*TP*CP*AP*GP*AP*CP*AP*CP*CP*AP*CP*TP...
+分子 #7: ZINC ION
+分子 #8: ADENOSINE-5'-DIPHOSPHATE
+分子 #9: BERYLLIUM TRIFLUORIDE ION
+分子 #10: MAGNESIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: UltrAuFoil R2/2 / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 3132 / 平均電子線量: 45.2 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 1.1 µm |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)