+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | DNA elongation complex (configuration 2) of Xenopus laevis DNA polymerase alpha-primase | ||||||||||||
 マップデータ マップデータ | DNA elongation complex (configuration 2) of Xenopus laevis DNA polymerase alpha-primase | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Primase / DNA polymerase / chimeric RNA-DNA primer / RNA/DNA hybrid / DNA replication / DNA synthesis / REPLICATION / TRANSFERASE-DNA-RNA complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alpha DNA polymerase:primase complex / DNA primase activity / primosome complex / DNA replication, synthesis of primer / lagging strand elongation / mitotic DNA replication initiation / DNA strand elongation involved in DNA replication / leading strand elongation / DNA replication initiation / DNA-directed RNA polymerase complex ...alpha DNA polymerase:primase complex / DNA primase activity / primosome complex / DNA replication, synthesis of primer / lagging strand elongation / mitotic DNA replication initiation / DNA strand elongation involved in DNA replication / leading strand elongation / DNA replication initiation / DNA-directed RNA polymerase complex / double-strand break repair via nonhomologous end joining / nuclear matrix / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / nuclear envelope / 4 iron, 4 sulfur cluster binding / DNA replication / DNA-directed DNA polymerase / DNA-directed DNA polymerase activity / nucleotide binding / chromatin binding / chromatin / nucleolus / DNA binding / nucleoplasm / nucleus / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 | |||||||||||||
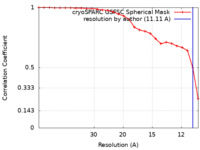
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.11 Å | ||||||||||||
 データ登録者 データ登録者 | Mullins EA / Durie CL / Ohi MD / Chazin WJ / Eichman BF | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: A mechanistic model of primer synthesis from catalytic structures of DNA polymerase α-primase. 著者: Elwood A Mullins / Lauren E Salay / Clarissa L Durie / Noah P Bradley / Jane E Jackman / Melanie D Ohi / Walter J Chazin / Brandt F Eichman /  要旨: The mechanism by which polymerase α-primase (polα-primase) synthesizes chimeric RNA-DNA primers of defined length and composition, necessary for replication fidelity and genome stability, is ...The mechanism by which polymerase α-primase (polα-primase) synthesizes chimeric RNA-DNA primers of defined length and composition, necessary for replication fidelity and genome stability, is unknown. Here, we report cryo-EM structures of Xenopus laevis polα-primase in complex with primed templates representing various stages of DNA synthesis. Our data show how interaction of the primase regulatory subunit with the primer 5' end facilitates handoff of the primer to polα and increases polα processivity, thereby regulating both RNA and DNA composition. The structures detail how flexibility within the heterotetramer enables synthesis across two active sites and provide evidence that termination of DNA synthesis is facilitated by reduction of polα and primase affinities for the varied conformations along the chimeric primer-template duplex. Together, these findings elucidate a critical catalytic step in replication initiation and provide a comprehensive model for primer synthesis by polα-primase. #1:  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: A mechanistic model of primer synthesis from catalytic structures of DNA polymerase alpha-primase 著者: Mullins EA / Salay LE / Durie CL / Bradley NP / Jackman JE / Ohi MD / Chazin WJ / Eichman BF | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42993.map.gz emd_42993.map.gz | 917 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42993-v30.xml emd-42993-v30.xml emd-42993.xml emd-42993.xml | 28.4 KB 28.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42993_fsc.xml emd_42993_fsc.xml | 2.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42993.png emd_42993.png | 25.7 KB | ||
| Filedesc metadata |  emd-42993.cif.gz emd-42993.cif.gz | 8.6 KB | ||
| その他 |  emd_42993_half_map_1.map.gz emd_42993_half_map_1.map.gz emd_42993_half_map_2.map.gz emd_42993_half_map_2.map.gz | 952.5 KB 952.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42993 http://ftp.pdbj.org/pub/emdb/structures/EMD-42993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42993 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42993_validation.pdf.gz emd_42993_validation.pdf.gz | 547.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42993_full_validation.pdf.gz emd_42993_full_validation.pdf.gz | 547.2 KB | 表示 | |
| XML形式データ |  emd_42993_validation.xml.gz emd_42993_validation.xml.gz | 7.9 KB | 表示 | |
| CIF形式データ |  emd_42993_validation.cif.gz emd_42993_validation.cif.gz | 9.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42993 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42993 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42993 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42993 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8v6jMC  8g99C  8g9fC  8g9lC  8g9nC  8g9oC  8ucuC  8ucvC  8ucwC  8v5mC  8v5nC  8v5oC  8v6gC  8v6hC  8v6iC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42993.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42993.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DNA elongation complex (configuration 2) of Xenopus laevis DNA polymerase alpha-primase | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.11875 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: DNA elongation complex (configuration 2) of Xenopus laevis...
| ファイル | emd_42993_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DNA elongation complex (configuration 2) of Xenopus laevis DNA polymerase alpha-primase (half 1) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: DNA elongation complex (configuration 2) of Xenopus laevis...
| ファイル | emd_42993_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DNA elongation complex (configuration 2) of Xenopus laevis DNA polymerase alpha-primase (half 2) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Polymerase alpha-primase with a DNA elongation substrate
+超分子 #1: Polymerase alpha-primase with a DNA elongation substrate
+超分子 #2: Polymerase alpha
+超分子 #3: Primase
+超分子 #4: DNA elongation substrate
+分子 #1: DNA polymerase alpha catalytic subunit
+分子 #2: DNA polymerase alpha subunit B
+分子 #3: DNA primase large subunit
+分子 #4: DNA primase
+分子 #5: DNA template
+分子 #6: RNA-DNA primer
+分子 #7: ZINC ION
+分子 #8: 2'-DEOXYGUANOSINE-5'-TRIPHOSPHATE
+分子 #9: MAGNESIUM ION
+分子 #10: IRON/SULFUR CLUSTER
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 3 / 実像数: 13641 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 45000 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)