+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The structure of the Clostridium thermocellum AdhE spirosome | |||||||||
 マップデータ マップデータ | Map of the AdhE spirosome | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Aldehyde-alcohol dehydrogenase / Complex / Spirosome / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methylglyoxal reductase (NADPH) (acetol producing) activity / butanol dehydrogenase (NAD+) activity / acetaldehyde dehydrogenase (acetylating) activity / alcohol metabolic process / alcohol dehydrogenase (NADP+) activity / carbon utilization / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Acetivibrio thermocellus DSM 1313 (バクテリア) Acetivibrio thermocellus DSM 1313 (バクテリア) | |||||||||
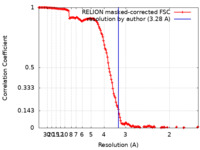
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.28 Å | |||||||||
 データ登録者 データ登録者 | Ziegler SJ / Gruber JN | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Structural characterization and dynamics of AdhE ultrastructures from Clostridium thermocellum: A containment strategy for toxic intermediates 著者: Ziegler S / Knott B / Gruber J / Hengge N / Xu Q / Olson D / Romero E / Joubert L / Bomble Y | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42284.map.gz emd_42284.map.gz | 249.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42284-v30.xml emd-42284-v30.xml emd-42284.xml emd-42284.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42284_fsc.xml emd_42284_fsc.xml | 14.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42284.png emd_42284.png | 74.1 KB | ||
| マスクデータ |  emd_42284_msk_1.map emd_42284_msk_1.map | 266.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42284.cif.gz emd-42284.cif.gz | 6.3 KB | ||
| その他 |  emd_42284_half_map_1.map.gz emd_42284_half_map_1.map.gz emd_42284_half_map_2.map.gz emd_42284_half_map_2.map.gz | 212.7 MB 213.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42284 http://ftp.pdbj.org/pub/emdb/structures/EMD-42284 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42284 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42284 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8uhwMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42284.map.gz / 形式: CCP4 / 大きさ: 266.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42284.map.gz / 形式: CCP4 / 大きさ: 266.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of the AdhE spirosome | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
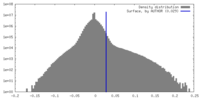
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42284_msk_1.map emd_42284_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
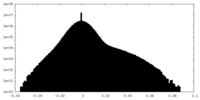
| 投影像・断面図 |
| ||||||||||||
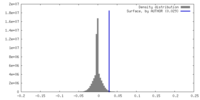
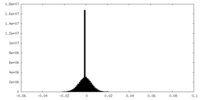
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_42284_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
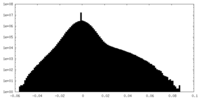
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_42284_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homo-oligomer complex of AdhE
| 全体 | 名称: Homo-oligomer complex of AdhE |
|---|---|
| 要素 |
|
-超分子 #1: Homo-oligomer complex of AdhE
| 超分子 | 名称: Homo-oligomer complex of AdhE / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Acetivibrio thermocellus DSM 1313 (バクテリア) Acetivibrio thermocellus DSM 1313 (バクテリア) |
-分子 #1: Aldehyde-alcohol dehydrogenase
| 分子 | 名称: Aldehyde-alcohol dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acetivibrio thermocellus DSM 1313 (バクテリア) Acetivibrio thermocellus DSM 1313 (バクテリア) |
| 分子量 | 理論値: 97.93868 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHENL YFQGMTKIAN KYEVIDNVEK LEKALKRLRE AQSVYATYTQ EQVDKIFFEA AMAANKMRIP LAKMAVEETG MGVVEDKVI KNHYASEYIY NAYKNTKTCG VIEEDPAFGI KKIAEPLGVI AAVIPTTNPT STAIFKTLIA LKTRNAIIIS P HPRAKNST ...文字列: MHHHHHHENL YFQGMTKIAN KYEVIDNVEK LEKALKRLRE AQSVYATYTQ EQVDKIFFEA AMAANKMRIP LAKMAVEETG MGVVEDKVI KNHYASEYIY NAYKNTKTCG VIEEDPAFGI KKIAEPLGVI AAVIPTTNPT STAIFKTLIA LKTRNAIIIS P HPRAKNST IEAAKIVLEA AVKAGAPEGI IGWIDVPSLE LTNLVMREAD VILATGGPGL VKAAYSSGKP AIGVGAGNTP AI IDDSADI VLAVNSIIHS KTFDNGMICA SEQSVIVLDG VYKEVKKEFE KRGCYFLNED ETEKVRKTII INGALNAKIV GQK AHTIAN LAGFEVPETT KILIGEVTSV DISEEFAHEK LCPVLAMYRA KDFDDALDKA ERLVADGGFG HTSSLYIDTV TQKE KLQKF SERMKTCRIL VNTPSSQGGI GDLYNFKLAP SLTLGCGSWG GNSVSDNVGV KHLLNIKTVA ERRENMLWFR TPEKI YIKR GCLPVALDEL KNVMGKKKAF IVTDNFLYNN GYTKPITDKL DEMGIVHKTF FDVSPDPSLA SAKAGAAEML AFQPDT IIA VGGGSAMDAA KIMWVMYEHP EVDFMDMAMR FMDIRKRVYT FPKMGQKAYF IAIPTSAGTG SEVTPFAVIT DEKTGIK YP LADYELLPDM AIVDADMMMN APKGLTAASG IDALTHALEA YVSMLATDYT DSLALRAIKM IFEYLPRAYE NGASDPVA R EKMANAATIA GMAFANAFLG VCHSMAHKLG AFYHLPHGVA NALMINEVIR FNSSEAPTKM GTFPQYDHPR TLERYAEIA DYIGLKGKNN EEKVENLIKA IDELKEKVGI RKTIKDYDID EKEFLDRLDE MVEQAFDDQC TGTNPRYPLM NEIRQMYLNA YYGGAKK UniProtKB: Aldehyde-alcohol dehydrogenase |
-分子 #2: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 2 / コピー数: 5 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 20 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8uhw: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)