+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Axle-less Bacillus sp. PS3 F1 ATPase mutant | |||||||||
 マップデータ マップデータ | Sharpened axle-less Bacillus PS3 ATPase map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATPase / ATP synthase / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proton motive force-driven plasma membrane ATP synthesis / proton-transporting ATP synthase complex, catalytic core F(1) / H+-transporting two-sector ATPase / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / ADP binding / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
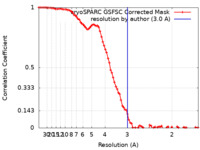
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Furlong EJ / Zeng YC / Brown SHJ / Sobti M / Stewart AG | |||||||||
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biochim Biophys Acta Bioenerg / 年: 2024 ジャーナル: Biochim Biophys Acta Bioenerg / 年: 2024タイトル: The molecular structure of an axle-less F-ATPase. 著者: Emily J Furlong / Ian-Blaine P Reininger-Chatzigiannakis / Yi C Zeng / Simon H J Brown / Meghna Sobti / Alastair G Stewart /  要旨: FF ATP synthase is a molecular rotary motor that can generate ATP using a transmembrane proton motive force. Isolated F-ATPase catalytic cores can hydrolyse ATP, passing through a series of ...FF ATP synthase is a molecular rotary motor that can generate ATP using a transmembrane proton motive force. Isolated F-ATPase catalytic cores can hydrolyse ATP, passing through a series of conformational states involving rotation of the central γ rotor subunit and the opening and closing of the catalytic β subunits. Cooperativity in F-ATPase has long thought to be conferred through the γ subunit, with three key interaction sites between the γ and β subunits being identified. Single molecule studies have demonstrated that the F complexes lacking the γ axle still "rotate" and hydrolyse ATP, but with less efficiency. We solved the cryogenic electron microscopy structure of an axle-less Bacillus sp. PS3 F-ATPase. The unexpected binding-dwell conformation of the structure in combination with the observed lack of interactions between the axle-less γ and the open β subunit suggests that the complete γ subunit is important for coordinating efficient ATP binding of F-ATPase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41811.map.gz emd_41811.map.gz | 56.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41811-v30.xml emd-41811-v30.xml emd-41811.xml emd-41811.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41811_fsc.xml emd_41811_fsc.xml | 8.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41811.png emd_41811.png | 57.1 KB | ||
| マスクデータ |  emd_41811_msk_1.map emd_41811_msk_1.map | 59.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41811.cif.gz emd-41811.cif.gz | 7 KB | ||
| その他 |  emd_41811_additional_1.map.gz emd_41811_additional_1.map.gz emd_41811_half_map_1.map.gz emd_41811_half_map_1.map.gz emd_41811_half_map_2.map.gz emd_41811_half_map_2.map.gz | 29.9 MB 55.4 MB 55.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41811 http://ftp.pdbj.org/pub/emdb/structures/EMD-41811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41811 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41811 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41811_validation.pdf.gz emd_41811_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41811_full_validation.pdf.gz emd_41811_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_41811_validation.xml.gz emd_41811_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_41811_validation.cif.gz emd_41811_validation.cif.gz | 20.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41811 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41811 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41811 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41811 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8u1hMC  9avjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41811.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41811.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened axle-less Bacillus PS3 ATPase map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
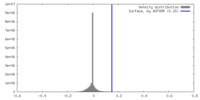
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41811_msk_1.map emd_41811_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
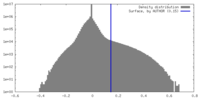
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bacillus PS3 F1 ATPase complex, with truncated gamma subunit
| 全体 | 名称: Bacillus PS3 F1 ATPase complex, with truncated gamma subunit |
|---|---|
| 要素 |
|
-超分子 #1: Bacillus PS3 F1 ATPase complex, with truncated gamma subunit
| 超分子 | 名称: Bacillus PS3 F1 ATPase complex, with truncated gamma subunit タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: The first 4 and last 25 amino acids have been deleted from the gamma subunit |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 354 KDa |
-分子 #1: ATP synthase subunit alpha
| 分子 | 名称: ATP synthase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 54.908676 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSIRAEEISA LIKQQIENYE SQIQVSDVGT VIQVGDGIAR AHGLDNVMSG ELVEFANGVM GMALNLEENN VGIVILGPYT GIKEGDEVR RTGRIMEVPV GEALIGRVVN PLGQPVDGLG PVETTETRPI ESRAPGVMDR RSVHEPLQTG IKAIDALVPI G RGQRELII ...文字列: MSIRAEEISA LIKQQIENYE SQIQVSDVGT VIQVGDGIAR AHGLDNVMSG ELVEFANGVM GMALNLEENN VGIVILGPYT GIKEGDEVR RTGRIMEVPV GEALIGRVVN PLGQPVDGLG PVETTETRPI ESRAPGVMDR RSVHEPLQTG IKAIDALVPI G RGQRELII GDRQTGKTSV AIDTIINQKD QNMISIYVAI GQKESTVRTV VETLRKHGAL DYTIVVTASA SQPAPLLFLA PY AGVAMGE YFMYKGKHVL VVYDDLSKQA AAYRELSLLL RRPPGREAYP GDIFYLHSRL LERAAKLSDA KGGGSLTALP FVE TQAGDI SAYIPTNVIS ITDGQIFLQS DLFFSGVRPA INAGLSVSRV GGAAQIKAMK KVAGTLRLDL AAYRELEAFA QFGS DLDKA TQAKLARGAR TVEVLKQDLH QPIPVEKQVL IIYALTRGFL DDIPVEDVRR FEKEFYLFLD QNGQHLLEHI RTTKD LPNE DDLNKAIEAF KKTFVVSQ UniProtKB: ATP synthase subunit alpha |
-分子 #2: ATP synthase subunit beta
| 分子 | 名称: ATP synthase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.424625 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHHHH HMTRGRVIQV MGPVVDVKFE NGHLPAIYNA LKIQHKARNE NEVDIDLTLE VALHLGDDTV RTIAMASTDG LIRGMEVID TGAPISVPVG EVTLGRVFNV LGEPIDLEGD IPADARRDPI HRPAPKFEEL ATEVEILETG IKVVDLLAPY I KGGKIGLF ...文字列: MHHHHHHHHH HMTRGRVIQV MGPVVDVKFE NGHLPAIYNA LKIQHKARNE NEVDIDLTLE VALHLGDDTV RTIAMASTDG LIRGMEVID TGAPISVPVG EVTLGRVFNV LGEPIDLEGD IPADARRDPI HRPAPKFEEL ATEVEILETG IKVVDLLAPY I KGGKIGLF GGAGVGKTVL IQELIHNIAQ EHGGISVFAG VGERTREGND LYHEMKDSGV ISKTAMVFGQ MNEPPGARMR VA LTGLTMA EYFRDEQGQD VLLFIDNIFR FTQAGSEVSA LLGRMPSAVG YQPTLATEMG QLQERITSTA KGSITSIQAI YVP ADDYTD PAPATTFSHL DATTNLERKL AEMGIYPAVD PLASTSRALA PEIVGEEHYQ VARKVQQTLQ RYKELQDIIA ILGM DELSD EDKLVVHRAR RIQFFLSQNF HVAEQFTGQP GSYVPVKETV RGFKEILEGK YDHLPEDAFR LVGRIEEVVE KAKAM GVEV UniProtKB: ATP synthase subunit beta |
-分子 #3: ATP synthase gamma chain
| 分子 | 名称: ATP synthase gamma chain / タイプ: protein_or_peptide / ID: 3 詳細: 4 residues deleted from N-term, 25 residues deleted from C-term, S109C, I212C, Strep-tag II insertion at N197 コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 29.677084 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDIKTRINAT KKTSQITKAM EMVSTSKLNR AEQNAKSFVP YMEKIQEVVA NVALGAGGAS HPMLVSRPVK KTGYLVITSD RGLAGAYNS NVLRLVYQTI QKRHACPDEY AIIVIGRVGL SFFRKRNMPV ILDITRLPDQ PSFADIKEIA RKTVGLFADG T FDELYMYY ...文字列: MDIKTRINAT KKTSQITKAM EMVSTSKLNR AEQNAKSFVP YMEKIQEVVA NVALGAGGAS HPMLVSRPVK KTGYLVITSD RGLAGAYNS NVLRLVYQTI QKRHACPDEY AIIVIGRVGL SFFRKRNMPV ILDITRLPDQ PSFADIKEIA RKTVGLFADG T FDELYMYY NHYVSAIQQE VTERKLLPLT DLAENWSHPQ FEKQRTVYEF EPSQEECLDV LLPQYAESLI YGALLDAKAS EH AARMTAM KNATDNANEL IRTLTL UniProtKB: ATP synthase gamma chain |
-分子 #4: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 4 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #7: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 7 / コピー数: 2 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL |
|---|---|
| 緩衝液 | pH: 7 詳細: 100 mM Potassium phosphate pH 7, 2 mM EDTA 1 mM AMPPNP was added 20 min prior to freezing |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR 詳細: Grid was glow discharged using a PELCO easiGlow set to 15 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 59.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | ModelAngelo was used to build an initial model into the map |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8u1h: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)