+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A mechanistic understanding of protective influenza B neuraminidase mAbs at the airway interface | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Neuraminidase Sialidase / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / : / exo-alpha-sialidase / carbohydrate metabolic process / host cell plasma membrane / virion membrane / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Influenza B virus (B/Iowa/06/2017) (B型インフルエンザウイルス) Influenza B virus (B/Iowa/06/2017) (B型インフルエンザウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.89 Å | |||||||||
 データ登録者 データ登録者 | Ferguson JA / Oeverdieck S / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: A mechanistic understanding of protective influenza B neuraminidase mAbs at the airway interface 著者: Ferguson JA / Oeverdieck S / Ward AB | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41809.map.gz emd_41809.map.gz | 483.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41809-v30.xml emd-41809-v30.xml emd-41809.xml emd-41809.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41809.png emd_41809.png | 40.8 KB | ||
| Filedesc metadata |  emd-41809.cif.gz emd-41809.cif.gz | 6.7 KB | ||
| その他 |  emd_41809_additional_1.map.gz emd_41809_additional_1.map.gz emd_41809_half_map_1.map.gz emd_41809_half_map_1.map.gz emd_41809_half_map_2.map.gz emd_41809_half_map_2.map.gz | 93 MB 474 MB 474 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41809 http://ftp.pdbj.org/pub/emdb/structures/EMD-41809 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41809 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41809 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41809_validation.pdf.gz emd_41809_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41809_full_validation.pdf.gz emd_41809_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_41809_validation.xml.gz emd_41809_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_41809_validation.cif.gz emd_41809_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41809 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41809 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41809 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41809 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41809.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41809.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.725 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Downsampled map used for Rosetta relax
| ファイル | emd_41809_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Downsampled map used for Rosetta_relax | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
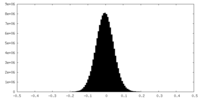
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41809_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
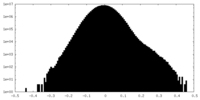
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41809_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of human donor mAb-fv domain bound to influenza B neurami...
| 全体 | 名称: Complex of human donor mAb-fv domain bound to influenza B neuraminidase |
|---|---|
| 要素 |
|
-超分子 #1: Complex of human donor mAb-fv domain bound to influenza B neurami...
| 超分子 | 名称: Complex of human donor mAb-fv domain bound to influenza B neuraminidase タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: mAb-400 heavy chain
| 分子 | 名称: mAb-400 heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.438196 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GLQLVQSGAE LKKPGESLKI SCQGSGYTFP IYWIGWVRQI PGKGLEWMGI IYPGDSDTRY SPSFEGQVTI SADKSVTTAY LQWSSLKAS DTAMYYCARH SEPWYPDHRF WPPYPFDIWG QGTMVTVSS |
-分子 #2: mAb-400 light chain
| 分子 | 名称: mAb-400 light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.529794 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EIVLTQSPGT LSLSPGERAT LSCRSSQTVS SGYLAWYQQK RGQAPRLLIY GASSRATGIP DRFSGSGSGT DFTLTISRLE PEDFAVYFC QHHGTSPGTF GGGTKVEIK |
-分子 #3: Neuraminidase
| 分子 | 名称: Neuraminidase / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza B virus (B/Iowa/06/2017) (B型インフルエンザウイルス) Influenza B virus (B/Iowa/06/2017) (B型インフルエンザウイルス) |
| 分子量 | 理論値: 51.104258 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLPSTIQTLT LFLTSGGVLL SLYVSASLSY LLYSDILLKF SPTEITAPTM PLDCANASNV QAVNRSATKG VTLLLPGPEW TYPRLSCPG STFQKALLIS PHRFGETKGN SAPLIIREPF VACGPNECKH FALTHYAAQP GGYYNGTRGD RNKLRHLISV K LGKIPTVE ...文字列: MLPSTIQTLT LFLTSGGVLL SLYVSASLSY LLYSDILLKF SPTEITAPTM PLDCANASNV QAVNRSATKG VTLLLPGPEW TYPRLSCPG STFQKALLIS PHRFGETKGN SAPLIIREPF VACGPNECKH FALTHYAAQP GGYYNGTRGD RNKLRHLISV K LGKIPTVE NSIFHMAAWS GSACHDGKEW TYIGVDGPDN NALLKVKYGE AYTDTYHSYA NNILRTQESA CNCIGGNCYL MI TDGSASG VSECRFLKIR EGRIIKEIFP TGRVKHTEEC TCGFASNKTI ECACRDNRYT AKRPFVKLNV ETDTAEIRLM CTD TYLDTP RPNDGSITGP CESDGDKGSG GIKGGFVHQR MKSKIGRWYS RTMSKTERMG MGLYVKYGGD PWADSDALAF SGVM VPMKE PGWYSFGFEI KDKKCDVPCI GIEMVHDGGK ETWHSAATAI YCLMGSGQLL WDTVTGVDMA L UniProtKB: Neuraminidase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: TBS |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 25 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4048 pixel / デジタル化 - サイズ - 縦: 4048 pixel / 撮影したグリッド数: 1 / 実像数: 5958 / 平均電子線量: 49.87 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 1.5 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 190000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 0.7000000000000001 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 190000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 得られたモデル |  PDB-8u1c: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)