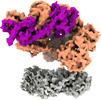
登録情報 データベース : EMDB / ID : EMD-41661タイトル Cryo-EM structure of S. cerevisiae Ctf18-RFC-PCNA complex in Apo state conformation I 複合体 : Ctf18-RFC-PCNA complexタンパク質・ペプチド : Replication factor C subunit 5タンパク質・ペプチド : Replication factor C subunit 4タンパク質・ペプチド : Replication factor C subunit 3タンパク質・ペプチド : Replication factor C subunit 2タンパク質・ペプチド : Chromosome transmission fidelity protein 18タンパク質・ペプチド : Proliferating cell nuclear antigenリガンド : ADENOSINE-5'-DIPHOSPHATEリガンド : PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTERリガンド : MAGNESIUM ION / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae (パン酵母)手法 / / 解像度 : 3.8 Å Yuan Z / Georgescu R / O'Donnell M / Li H 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) GM131754 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) GM115809 Howard Hughes Medical Institute (HHMI) M.E.O.
ジャーナル : Science / 年 : 2024タイトル : Mechanism of PCNA loading by Ctf18-RFC for leading-strand DNA synthesis.著者 : Zuanning Yuan / Roxana Georgescu / Nina Y Yao / Olga Yurieva / Michael E O'Donnell / Huilin Li / 要旨 : The proliferating cell nuclear antigen (PCNA) clamp encircles DNA to hold DNA polymerases (Pols) to DNA for processivity. The Ctf18-RFC PCNA loader, a replication factor C (RFC) variant, is specific ... The proliferating cell nuclear antigen (PCNA) clamp encircles DNA to hold DNA polymerases (Pols) to DNA for processivity. The Ctf18-RFC PCNA loader, a replication factor C (RFC) variant, is specific to the leading-strand Pol (Polε). We reveal here the underlying mechanism of Ctf18-RFC specificity to Polε using cryo-electron microscopy and biochemical studies. We found that both Ctf18-RFC and Polε contain specific structural features that direct PCNA loading onto DNA. Unlike other clamp loaders, Ctf18-RFC has a disordered ATPase associated with a diverse cellular activities (AAA+) motor that requires Polε to bind and stabilize it for efficient PCNA loading. In addition, Ctf18-RFC can pry prebound Polε off of DNA, then load PCNA onto DNA and transfer the PCNA-DNA back to Polε. These elements in both Ctf18-RFC and Polε provide specificity in loading PCNA onto DNA for Polε. 履歴 登録 2023年8月20日 - ヘッダ(付随情報) 公開 2024年9月4日 - マップ公開 2024年9月4日 - 更新 2024年10月9日 - 現状 2024年10月9日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Science / 年: 2024
ジャーナル: Science / 年: 2024
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_41661.map.gz
emd_41661.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-41661-v30.xml
emd-41661-v30.xml emd-41661.xml
emd-41661.xml EMDBヘッダ
EMDBヘッダ emd_41661.png
emd_41661.png emd-41661.cif.gz
emd-41661.cif.gz emd_41661_half_map_1.map.gz
emd_41661_half_map_1.map.gz emd_41661_half_map_2.map.gz
emd_41661_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-41661
http://ftp.pdbj.org/pub/emdb/structures/EMD-41661 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41661
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41661 emd_41661_validation.pdf.gz
emd_41661_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_41661_full_validation.pdf.gz
emd_41661_full_validation.pdf.gz emd_41661_validation.xml.gz
emd_41661_validation.xml.gz emd_41661_validation.cif.gz
emd_41661_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41661
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41661 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41661
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41661

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
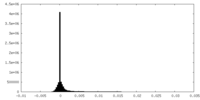
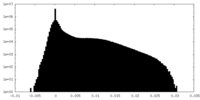
マップ ダウンロード / ファイル: emd_41661.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_41661.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)